Bezkomórkowe DNA płodu - Cell-free fetal DNA
Bezkomórkowy płodowy DNA ( cffDNA ) to płodowy DNA krążący swobodnie we krwi matki . Krew matki jest pobierana przez nakłucie żyły . Analiza cffDNA jest metodą nieinwazyjnej diagnostyki prenatalnej, często zlecaną kobietom ciężarnym w zaawansowanym wieku matki . Dwie godziny po porodzie cffDNA nie jest już wykrywalne we krwi matki.
Tło
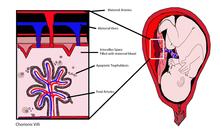
cffDNA pochodzi z trofoblastów łożyskowych . Płodowego DNA jest podzielona przy łożysku mikrocząstki są wydalane do matczyną krwi obiegu .
Fragmenty cffDNA mają długość około 200 par zasad (pz). Są znacznie mniejsze niż fragmenty DNA matki. Różnica w wielkości pozwala na odróżnienie cffDNA od matczynych fragmentów DNA.
Około 11 do 13,4% wolnego od komórek DNA we krwi matki jest pochodzenia płodowego. Kwota jest bardzo zróżnicowana w zależności od kobiety w ciąży. cffDNA jest obecne po pięciu do siedmiu tygodniach ciąży. Ilość cffDNA wzrasta wraz z postępem ciąży. Ilość cffDNA we krwi matki szybko spada po porodzie. Dwie godziny po porodzie cffDNA nie jest już wykrywalne we krwi matki.
Analiza cffDNA może zapewnić wcześniejszą diagnozę stanu płodu niż obecne techniki. Ponieważ cffDNA znajduje się we krwi matki, pobieranie próbek nie niesie ze sobą ryzyka samoistnego poronienia . Analiza cffDNA wiąże się z tymi samymi problemami etycznymi i praktycznymi, co inne techniki, takie jak amniopunkcja i biopsja kosmówki .
Niektóre wady pobierania próbek cffDNA obejmują niskie stężenie cffDNA we krwi matki; zmienność ilości cffDNA między osobnikami; wysokie stężenie DNA wolnego od komórek matczynych w porównaniu z cffDNA we krwi matki.
Nowe dowody wskazują, że odsetek niepowodzeń testu cffDNA jest wyższy, frakcja płodowa (proporcja DNA płodowego w porównaniu z matczynym DNA w próbce krwi matki) jest niższa, a PPV dla trisomii 18, 13 i SCA jest mniejsze w ciążach IVF w porównaniu z ciążami poczętymi spontanicznie.
Metody laboratoryjne
Opracowano szereg metod laboratoryjnych do bezkomórkowego badania przesiewowego DNA płodowego pod kątem defektów genetycznych. Główne z nich to (1) masowo równoległe sekwencjonowanie typu shotgun (MPSS), (2) ukierunkowane masowe sekwencjonowanie równoległe (t-MPS) oraz (3) podejście oparte na polimorfizmie pojedynczego nukleotydu (SNP).
Próbka krwi obwodowej matki jest pobierana przez weneksję około dziesięciu tygodni ciąży.
Separacja cffDNA
Osocze krwi jest oddzielane od próbki krwi matki za pomocą wirówki laboratoryjnej . Następnie cffDNA izoluje się i oczyszcza. Na podstawie oceny literatury naukowej sporządzono znormalizowany protokół wykonania tego zadania . Najwyższą wydajność ekstrakcji cffDNA uzyskano przy użyciu zestawu „QIAamp DSP Virus Kit”.
Dodatek formaldehydu do próbek krwi matki zwiększa wydajność cffDNA. Formaldehyd stabilizuje nienaruszone komórki, a zatem hamuje dalsze uwalnianie matczynego DNA. Po dodaniu formaldehydu procent cffDNA odzyskanego z próbki krwi matki waha się od 0,32 do 40 procent ze średnią 7,7 procent. Bez dodatku formaldehydu zmierzono średni procent odzyskanego cffDNA na 20,2%. Jednak inne liczby wahają się od 5 do 96 procent.
Odzyskiwanie cffDNA może być związane z długością fragmentów DNA. Inny sposób na zwiększenie DNA płodu opiera się na fizycznej długości fragmentów DNA. Mniejsze fragmenty mogą stanowić do siedemdziesięciu procent całkowitego wolnego od komórek DNA w próbce krwi matki.
Analiza cffDNA
W PCR w czasie rzeczywistym do monitorowania akumulacji amplikonów stosuje się sondy fluorescencyjne . Sygnał fluorescencyjny reportera jest proporcjonalny do liczby wytworzonych amplikonów. Najbardziej odpowiedni protokół PCR w czasie rzeczywistym jest zaprojektowany zgodnie z konkretną mutacją lub genotypem, który ma być wykryty. Mutacje punktowe są analizowane za pomocą jakościowej reakcji PCR w czasie rzeczywistym z użyciem sond specyficznych dla alleli . insercje i delecje są analizowane przez pomiary dawki przy użyciu ilościowego PCR w czasie rzeczywistym.
cffDNA można wykryć, znajdując sekwencje DNA odziedziczone po ojcu za pomocą reakcji łańcuchowej polimerazy (PCR).
Ilościowy PCR w czasie rzeczywistym
Gen determinujący płeć regionu Y (SRY) i krótkie powtórzenie tandemowe chromosomu Y „DYS14” w cffDNA z 511 ciąż analizowano za pomocą ilościowej reakcji PCR w czasie rzeczywistym (RT-qPCR). W 401 z 403 ciąż, w których pobrano krew matczyną w siódmym tygodniu ciąży lub więcej, znaleziono oba segmenty DNA.
Zagnieżdżony PCR
Oceniono zastosowanie zagnieżdżonej reakcji łańcuchowej polimerazy (nested PCR) w celu określenia płci poprzez wykrycie sygnału specyficznego dla chromosomu Y w cffDNA z osocza matki. Nested PCR wykrył 53 z 55 płodów płci męskiej. CffDNA z osocza 3 z 25 kobiet z płodami żeńskimi zawierał sygnał specyficzny dla chromosomu Y. Wrażliwość zagnieżdżonych PCR, w tym eksperymencie wynosiła 96 procent. Specyficzność wynosiła 88 procent.
Cyfrowy PCR
Urządzenia mikroprzepływowe umożliwiają ilościowe oznaczenie segmentów cffDNA w osoczu matki z dokładnością wykraczającą poza real-time PCR. Mutacje punktowe , utratę heterozygotyczności i aneuploidię można wykryć w jednym etapie PCR. Cyfrowy PCR umożliwia rozróżnienie między osoczem krwi matki a DNA płodu w sposób multipleksowy .
Sekwencjonowanie strzelb
Wysokowydajne sekwencjonowanie przy użyciu strzelby przy użyciu narzędzi takich jak Solexa lub Illumina daje około 5 milionów znaczników sekwencji na próbkę surowicy matki. Ciąże aneuploidalne, takie jak trisomia, zostały zidentyfikowane podczas badań w czternastym tygodniu ciąży. Mapowanie całego genomu płodu przez analizę haplotypów rodzicielskich zakończono sekwencjonowaniem cffDNA z surowicy matki. Ciężarne kobiety badano przy użyciu sekwencjonowania 2-pleksowego masowo równoległego DNA w osoczu matki i zdiagnozowano trisomię z wynikiem z większym niż 3. Sekwencjonowanie dało czułość 100 procent, swoistość 97,9 procent, pozytywną wartość predykcyjną 96,6 i negatywną wartość predykcyjna 100 procent.
Spekrtometria masy
Desorpcja/jonizacja laserowa wspomagana matrycą - spektrometria masowa czasu przelotu (MALDI-TOF MS) w połączeniu z wydłużaniem pojedynczych zasad po reakcji PCR umożliwia detekcję cffDNA ze specyficznością pojedynczej zasady i czułością pojedynczej cząsteczki DNA. DNA jest amplifikowane przez PCR. Następnie liniowa amplifikacja z reakcją wydłużania zasady (z trzecim starterem) jest zaprojektowana do przyłączenia do regionu powyżej miejsca mutacji . Do startera wydłużającego dodaje się jedną lub dwie zasady w celu wytworzenia dwóch produktów wydłużania z DNA typu dzikiego i zmutowanego DNA. Specyficzność pojedynczej zasady zapewnia przewagę nad technikami opartymi na hybrydyzacji z użyciem sond hydrolizy TaqMan . Podczas oceny tej techniki nie znaleziono wyników fałszywie dodatnich ani ujemnych podczas poszukiwania cffDNA w celu określenia płci płodu w szesnastu próbkach osocza matki. Płeć dziewięćdziesięciu jeden płodów płci męskiej została prawidłowo wykryta za pomocą spektrometrii masowej MALDI-TOF. Technika miała dokładność, czułość i swoistość ponad 99 procent.
Modyfikacje epigenetyczne
Można wykorzystać różnice w aktywacji genów między DNA matki i płodu. Do wykrywania cffDNA można zastosować modyfikacje epigenetyczne (dziedziczne modyfikacje, które zmieniają funkcję genu bez zmiany sekwencji DNA). Hipermetylowany RASSF1 Promotor jest uniwersalnym znacznik płodów w celu potwierdzenia obecności cffDNA. Opisano technikę, w której cffDNA ekstrahowano z osocza matki, a następnie trawiono enzymami restrykcyjnymi wrażliwymi i niewrażliwymi na metylację . Następnie przeprowadzono analizę PCR w czasie rzeczywistym RASSF1A, SRY i DYS14. Procedura wykryła 79 z 90 (88 procent) próbek krwi matki, w których obecny był hipermetylowany RASSF1A.
mRNA
Transkrypty mRNA z genów ulegających ekspresji w łożysku są wykrywalne w osoczu matki. W tej procedurze osocze jest odwirowywane, więc pojawia się warstwa wodna. Ta warstwa jest przenoszona iz niej ekstrahowany jest RNA . RT-PCR służy do wykrywania wybranej ekspresji RNA. Na przykład, ludzki laktogen łożyskowy (hPL) i mRNA beta-hCG są stabilne w osoczu matki i można je wykryć. (Ng i wsp. 2002). Może to pomóc w potwierdzeniu obecności cffDNA w osoczu matki.
Aplikacje
Prenatalne rozeznanie płci
Analiza cffDNA z próbki osocza matki pozwala na prenatalne rozeznanie płci . Zastosowania prenatalnego rozeznania płci obejmują:
- Badanie choroby : To, czy płeć płodu jest męska czy żeńska, pozwala określić ryzyko określonego recesywnego zaburzenia genetycznego sprzężonego z chromosomem X w konkretnej ciąży, zwłaszcza gdy matka jest genetycznym nosicielem tego zaburzenia.
- Przygotowanie do wszelkich aspektów rodzicielstwa zależnych od płci.
- Selekcja płci , która po preimplantacyjnej diagnostyce genetycznej może być wykonana poprzez selekcję tylko zarodków preferowanej płci, lub po metodach postimplantacyjnych poprzez dokonanie aborcji selekcyjnej w zależności od wyniku badania i osobistych preferencji.
W porównaniu z ultrasonografią położniczą, która nie jest wiarygodna w określaniu płci w I trymestrze ciąży i amniopunkcją, która niesie ze sobą niewielkie ryzyko poronienia , pobieranie próbek osocza matki do analizy cffDNA jest bez ryzyka. Głównymi celami analizy cffDNA są gen odpowiedzialny za białko regionu Y determinującego płeć (SRY) na chromosomie Y oraz sekwencja DYS14.
Wrodzony przerost nadnerczy
W przypadku wrodzonego przerostu nadnerczy, kora nadnerczy nie posiada odpowiedniej syntezy kortykosteroidów, co prowadzi do nadmiaru androgenów nadnerczowych i wpływa na płody żeńskie. U płodów żeńskich występuje zewnętrzna maskulinizacja genitaliów. Matki płodów z grupy ryzyka otrzymują deksametazon w 6. tygodniu ciąży w celu zahamowania uwalniania androgenów przez przysadkę mózgową .
Jeżeli analiza cffDNA uzyskanego z próbki osocza matki nie zawiera markerów genetycznych znajdujących się tylko na chromosomie Y, sugeruje to żeński płód. Może to jednak również wskazywać na niepowodzenie samej analizy (wynik fałszywie ujemny). Do wykrywania cffDNA można wykorzystać ojcowskie polimorfizmy genetyczne i markery niezależne od płci. W tym zastosowaniu musi być obecny wysoki stopień heterozygotyczności tych markerów.
Badanie ojcostwa
Testy prenatalnego DNA na ojcostwo są dostępne w handlu. Test można wykonać w dziewiątym tygodniu ciąży.
Zaburzenia pojedynczego genu
Autosomalne dominujące i recesywne zaburzenia jednogenowe, które zostały zdiagnozowane prenatalnie na podstawie analizy DNA dziedziczonego po ojcu, obejmują mukowiscydozę , talasemię beta , anemię sierpowatą , rdzeniowy zanik mięśni i dystrofię miotoniczną . Diagnostyka prenatalna zaburzeń pojedynczego genu, które są spowodowane autosomalną mutacją recesywną, mutacją autosomalną dominującą dziedziczoną przez matkę lub mutacją dużych sekwencji, która obejmuje duplikację, ekspansję lub insercję sekwencji DNA jest trudniejsza.
W cffDNA trudniej jest wykryć fragmenty o długości 200 – 300 pz związane z zaburzeniami pojedynczego genu.
Na przykład, stan autosomalny dominujący, achondroplazja, jest spowodowany mutacją punktową genu FGFR3. W dwóch ciążach płodu z achondroplazją stwierdzono mutację G1138A odziedziczoną po ojcu z cffDNA z próbki osocza matki w jednej i mutację G1138A de novo w drugiej.
W badaniach genetyki pląsawicy Huntingtona przy użyciu qRT-PCR cffDNA z próbek osocza matki, powtórzenia CAG wykryto na normalnych poziomach (17, 20 i 24).
cffDNA można również wykorzystać do diagnozowania zaburzeń pojedynczego genu . Zmiany w procesach laboratorium przy użyciu cffDNA może pozwolić prenatalnej diagnostyki z aneuploidii jak trisomią 21 (zespół Downa) u płodu.
Choroba hemolityczna płodu i noworodka
Niezgodność płodowych i matczynych antygenów RhD jest główną przyczyną choroby hemolitycznej noworodka . Około 15 procent kobiet rasy kaukaskiej , 3 do 5 procent czarnoskórych kobiet z Afryki i mniej niż 3% kobiet z Azji ma RhD-ujemny.
Dokładna diagnoza prenatalna jest ważna, ponieważ choroba może być śmiertelna dla noworodka, a matkom z grupy ryzyka można podawać domięśniową immunoglobulinę (anty-D) lub immunoglobulinę dożylną .
PCR do wykrywania RHD (gen) dla genu egzonów 5 i 7 z cffDNA uzyskaną z osocza matki od 9 do 13 tygodni ciąży daje wysoką specyficzność, czułość i dokładność diagnostyczną (> 90 procent) w porównaniu z RhD określenia od noworodków krwi pępowinowej surowicy . Podobne wyniki uzyskano w przypadku eksonów 7 i 10. Cyfrowa reakcja PCR z kroplami w oznaczaniu płodowego RhD była porównywalna z rutynową techniką PCR w czasie rzeczywistym.
Rutynowe oznaczanie stanu RhD płodu na podstawie cffDNA w surowicy matki pozwala na wczesne zarządzanie ciążami z ryzykiem przy jednoczesnym zmniejszeniu niepotrzebnego stosowania anty-D o ponad 25 procent.
Aneuploidia
- Chromosomy płciowe
Analiza surowicy matki cffDNA wysokotemperaturowego sekwencjonowania wykrywa wspólne płodu chromosomów płciowych aneuploidii , takich jak syndrom Turnera , zespół Klinefeltera i potrójnym zespołem X. a procedura jest dodatnia wartość predykcyjna jest niska.
- Trisomia 21
Trisomia płodu chromosomu 21 jest przyczyną zespołu Downa. Tę trisomię można wykryć poprzez analizę cffDNA z krwi matki za pomocą masowo równoległego sekwencjonowania shotgun (MPSS). Inną techniką jest analiza cyfrowa wybranych regionów (DANSR). Takie testy wykazują czułość około 99% i swoistość ponad 99,9%. Dlatego nie można ich traktować jako procedur diagnostycznych, ale można je wykorzystać do potwierdzenia pozytywnego wyniku matczynego badania przesiewowego, takiego jak badanie przesiewowe w pierwszym trymestrze lub ultrasonograficzne markery choroby.
- Trisomia 13 i 18
Możliwa jest analiza cffDNA z osocza matki za pomocą MPSS w poszukiwaniu trisomii 13 lub 18
Czynniki ograniczające czułość i swoistość obejmują poziomy cffDNA w osoczu matki; chromosomy matczyne mogą mieć mozaicyzm .
Można wykryć wiele płodowych cząsteczek kwasu nukleinowego pochodzących z chromosomów aneuploidalnych, w tym mRNA SERPINEB2, opłaszczony B, hipometylowany SERPINB5 z chromosomu 18, swoisty dla łożyska 4 (PLAC4), hipermetylowaną syntetazę holokarboksylazy (HLCS) i mRNA c21orf105 z chromosomu 12. trisomii, allele mRNA w osoczu matki nie są w normalnym stosunku 1:1, ale w rzeczywistości są to 2:1. Stosunki alleli określone przez markery epigenetyczne można również wykorzystać do wykrycia kompletnych trisomii. Masywne sekwencjonowanie równoległe i cyfrowy PCR do wykrywania aneuploidii płodu można stosować bez ograniczeń do cząsteczek kwasu nukleinowego swoistych dla płodu. Szacuje się, że (MPSS) ma czułość od 96 do 100%, a swoistość od 94 do 100% w wykrywaniu zespołu Downa. Można go wykonać w 10 tygodniu ciąży . W jednym badaniu w Stanach Zjednoczonych oszacowano odsetek wyników fałszywie dodatnich na 0,3% i dodatnią wartość predykcyjną na 80% przy użyciu cffDNA do wykrywania zespołu Downa.
Stan przedrzucawkowy
Stan przedrzucawkowy jest złożonym stanem ciąży obejmującym nadciśnienie i białkomocz, zwykle po 20. tygodniu ciąży. Wiąże się to ze słabą inwazją cytotrofoblastyczną mięśniówki macicy . Początek choroby między 20 a 34 tygodniem ciąży jest uważany za „wczesny”. Próbki osocza matki w ciążach powikłanych stanem przedrzucawkowym mają znacznie wyższe poziomy cffDNA niż w ciążach prawidłowych. Dotyczy to stanu przedrzucawkowego o wczesnym początku.
Perspektywy na przyszłość
Sekwencjonowanie nowej generacji może być wykorzystane do uzyskania sekwencji całego genomu z cffDNA. Rodzi to pytania etyczne. Jednak użyteczność procedury może wzrosnąć, gdy odkryte zostaną wyraźne powiązania między określonymi wariantami genetycznymi a stanami chorobowymi.