Synteza DNA - DNA synthesis
Synteza DNA to naturalne lub sztuczne tworzenie cząsteczek kwasu dezoksyrybonukleinowego (DNA). DNA to makrocząsteczka składająca się z jednostek nukleotydowych , które są połączone wiązaniami kowalencyjnymi i wodorowymi w powtarzającej się strukturze. Synteza DNA zachodzi, gdy te jednostki nukleotydowe są połączone, tworząc DNA; może to nastąpić sztucznie ( in vitro ) lub naturalnie ( in vivo ). Jednostki nukleotydowe składają się z zasady azotowej (cytozyny, guaniny, adeniny lub tyminy), cukru pentozowego (dezoksyrybozy) i grupy fosforanowej. Każda jednostka jest połączona, gdy między jej grupą fosforanową a cukrem pentozowym następnego nukleotydu tworzy się wiązanie kowalencyjne, tworząc szkielet cukier-fosforan. DNA jest komplementarną, dwuniciową strukturą, ponieważ specyficzne parowanie zasad (adenina i tymina, guanina i cytozyna) występuje naturalnie, gdy wiązania wodorowe tworzą się między zasadami nukleotydowymi.
Istnieje kilka różnych definicji syntezy DNA: może odnosić się do replikacji DNA - biosyntezy DNA ( amplifikacja DNA in vivo ), reakcji łańcuchowej polimerazy - enzymatycznej syntezy DNA ( amplifikacja DNA in vitro ) lub syntezy genów - fizycznego tworzenia sztucznych sekwencji genów . Chociaż każdy rodzaj syntezy jest bardzo inny, mają one pewne cechy. Nukleotydy, które zostały połączone w polinukleotydy, mogą działać jako matryca DNA dla jednej formy syntezy DNA – PCR – zachodzącej. Replikacja DNA działa również przy użyciu matrycy DNA, podwójna helisa DNA rozwija się podczas replikacji, odsłaniając niesparowane zasady dla nowych nukleotydów na wiązanie wodorowe. Synteza genów nie wymaga jednak matrycy DNA, a geny są składane de novo .
Synteza DNA zachodzi u wszystkich eukariontów i prokariontów , a także u niektórych wirusów . Dokładna synteza DNA jest ważna, aby uniknąć mutacji DNA. U ludzi mutacje mogą prowadzić do chorób, takich jak rak, więc synteza DNA i mechanizmy zaangażowane in vivo były intensywnie badane przez dziesięciolecia. W przyszłości badania te mogą posłużyć do opracowania technologii związanych z syntezą DNA do wykorzystania w przechowywaniu danych.
replikacja DNA
W naturze cząsteczki DNA są syntetyzowane przez wszystkie żywe komórki w procesie replikacji DNA. Zwykle dzieje się to jako część podziału komórki. Replikacja DNA zachodzi, więc podczas podziału komórki każda komórka potomna zawiera dokładną kopię materiału genetycznego komórki. Synteza DNA in vivo (replikacja DNA) zależy od złożonego zestawu enzymów, które wyewoluowały, aby działać w skoordynowany sposób podczas fazy S cyklu komórkowego. Zarówno u eukariontów, jak i prokariontów replikacja DNA zachodzi, gdy specyficzne topoizomerazy, helikazy i gyrazy (białka inicjujące replikację) rozpinają dwuniciowy DNA, odsłaniając zasady azotowe. Enzymy te wraz z białkami pomocniczymi tworzą makromolekularną maszynę, która zapewnia dokładną duplikację sekwencji DNA. Następuje komplementarne parowanie zasad, tworząc nową dwuniciową cząsteczkę DNA. Jest to znane jako Semikonserwatywna replikacji od jednej nici nową cząsteczkę DNA pochodzi z macierzystymi części.
W sposób ciągły enzymy eukariotyczne napotykają uszkodzenia DNA, które mogą zaburzać replikację DNA. Uszkodzenie to ma postać uszkodzeń DNA, które powstają samoistnie lub z powodu czynników uszkadzających DNA. Maszyneria replikacji DNA jest zatem ściśle kontrolowana, aby zapobiec zapadaniu się w przypadku napotkania uszkodzenia. Kontrola systemu replikacji DNA zapewnia replikację genomu tylko raz na cykl; nadmierna replikacja powoduje uszkodzenie DNA. Deregulacja replikacji DNA jest kluczowym czynnikiem niestabilności genomu podczas rozwoju raka.
Podkreśla to specyfikę maszynerii syntezy DNA in vivo . Istnieją różne sposoby sztucznego stymulowania replikacji naturalnie występującego DNA lub tworzenia sztucznych sekwencji genów. Jednak synteza DNA in vitro może być procesem bardzo podatnym na błędy.
Synteza naprawy DNA
Uszkodzone DNA podlega naprawie w kilku różnych procesach enzymatycznych , w których każdy indywidualny proces jest wyspecjalizowany w naprawie określonych rodzajów uszkodzeń. DNA człowieka ulega uszkodzeniu z wielu źródeł naturalnych, a niewystarczająca naprawa jest związana z chorobami i przedwczesnym starzeniem się . Większość procesów naprawy DNA tworzy jednoniciowe luki w DNA podczas pośredniego etapu naprawy, a luki te są wypełniane przez syntezę naprawczą. Specyficzne procesy naprawcze, które wymagają wypełniania szczelin poprzez syntezę DNA obejmują nukleotydów excision repair , naprawa przez wycinanie zasady , naprawy błędnie sparowanych zasad , homologicznej recombinational naprawy, niehomologicznej końcówkę łączącą i koniec microhomology pośredniczy łączącą .
Transkrypcja odwrotna
Odwrotna transkrypcja jest częścią cyklu replikacji poszczególnych rodzin wirusów, w tym retrowirusów . Polega na kopiowaniu RNA do dwuniciowego komplementarnego DNA (cDNA) przy użyciu enzymów odwrotnej transkryptazy . W retrowirusach wirusowy RNA jest wstawiany do jądra komórki gospodarza. Tam wirusowy enzym odwrotnej transkryptazy dodaje nukleotydy DNA do sekwencji RNA, tworząc cDNA, który jest wstawiany do genomu komórki gospodarza przez enzym integrazę kodującą białka wirusowe.
Reakcja łańcuchowa polimerazy
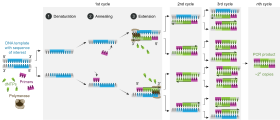
Łańcuchowa reakcja polimerazy jest formą enzymatycznej syntezy DNA w warunkach laboratoryjnych za pomocą powtarzających się cykli grzania i chłodzenia w reakcji do topienia DNA i enzymatyczne replikacji DNA.
Synteza DNA podczas PCR jest bardzo podobna do żywych komórek, ale ma bardzo specyficzne odczynniki i warunki. Podczas PCR DNA jest chemicznie ekstrahowane z białek opiekuńczych gospodarza, a następnie podgrzewane, co powoduje termiczną dysocjację nici DNA. Z oryginalnej nici zbudowane są dwie nowe nici cDNA, które można ponownie podzielić, aby działały jako matryca dla dalszych produktów PCR. Oryginalne DNA jest mnożone przez wiele rund PCR. Można wykonać ponad miliard kopii oryginalnej nici DNA.
Losowa mutageneza
W przypadku wielu eksperymentów, takich jak badania strukturalne i ewolucyjne, naukowcy muszą stworzyć dużą bibliotekę wariantów określonej sekwencji DNA. Mutageneza losowa ma miejsce in vitro, gdy mutagenna replikacja z polimerazą DNA o niskiej wierności jest połączona z selektywną amplifikacją PCR w celu wytworzenia wielu kopii zmutowanego DNA.
RT-PCR
RT-PCR różni się od konwencjonalnego PCR, ponieważ syntetyzuje cDNA z mRNA, a nie matrycowego DNA. Technika ta łączy reakcję odwrotnej transkrypcji z amplifikacją opartą na PCR, ponieważ sekwencja RNA działa jako matryca dla enzymu, odwrotnej transkryptazy. RT-PCR jest często stosowany do badania ekspresji genów w określonych typach tkanek lub komórek na różnych etapach rozwoju lub do badania zaburzeń genetycznych.
Synteza genów
Sztuczna synteza genów to proces syntezy genu in vitro bez potrzeby pobierania początkowych próbek DNA matrycy. W 2010 roku J. Craig Venter i jego zespół jako pierwsi wykorzystali całkowicie zsyntetyzowane DNA do stworzenia samoreplikującego się mikroba, nazwanego laboratorium Mycoplasma .
Synteza oligonukleotydów
Synteza oligonukleotydów to chemiczna synteza sekwencji kwasów nukleinowych. Większość badań biologicznych i bioinżynierii dotyczy syntetycznego DNA, które może zawierać oligonukleotydy , syntetyczne geny, a nawet chromosomy . Obecnie całe syntetyczne DNA jest budowane na zamówienie metodą amidofosforynową przez Marvina H. Caruthersa . Oligo są syntetyzowane z cegiełek, które są repliką naturalnych zasad. Proces jest zautomatyzowany od późnych lat 70. i może być wykorzystywany do tworzenia pożądanych sekwencji genetycznych, a także do innych zastosowań w medycynie i biologii molekularnej. Jednak chemiczne tworzenie sekwencji jest niepraktyczne powyżej 200-300 zasad i jest procesem niebezpiecznym dla środowiska. Te oligonukleotydy, liczące około 200 zasad, można łączyć metodami składania DNA, tworząc większe cząsteczki DNA.
W niektórych badaniach zbadano możliwość syntezy enzymatycznej przy użyciu terminalnej transferazy deoksynukleotydylowej (TdT), polimerazy DNA, która nie wymaga matrycy. Jednak ta metoda nie jest jeszcze tak skuteczna jak synteza chemiczna i nie jest dostępna na rynku.
Wraz z postępami w sztucznej syntezie DNA badana jest możliwość przechowywania danych DNA . Dzięki ultrawysokiej gęstości przechowywania i długoterminowej stabilności, syntetyczne DNA jest interesującą opcją do przechowywania dużych ilości danych. Chociaż informacje można bardzo szybko uzyskać z DNA za pomocą technologii sekwencjonowania nowej generacji, synteza DNA de novo jest głównym wąskim gardłem w tym procesie. Na cykl można dodać tylko jeden nukleotyd, a każdy cykl zajmuje kilka sekund, więc ogólna synteza jest bardzo czasochłonna i bardzo podatna na błędy. Jeśli jednak biotechnologia ulegnie poprawie, syntetyczne DNA może pewnego dnia zostać wykorzystane do przechowywania danych.
Synteza par zasad
Istnieją doniesienia, że mogą być syntetyzowane nowe pary nukleozasad , a także AT ( adenina - tymina ) i GC ( guanina - cytozyna ). Syntetyczne nukleotydy mogą być użyte do rozszerzenia alfabetu genetycznego i umożliwienia specyficznej modyfikacji miejsc DNA. Nawet tylko trzecia para zasad rozszerzyłaby liczbę aminokwasów, które mogą być kodowane przez DNA, z istniejących 20 aminokwasów do możliwych 172. DNA Hachimoji jest zbudowany z ośmiu liter nukleotydów, tworząc cztery możliwe pary zasad. Jest to zatem podwójna gęstość informacji naturalnego DNA. W badaniach RNA zostało nawet wyprodukowane z DNA hachimoji. Ta technologia może być również wykorzystana do przechowywania danych w DNA.