Projekt bazy danych genomu Ensemble - Ensembl genome database project
 | |
|---|---|
| Zadowolony | |
| Opis | Zespół |
| Kontakt | |
| Centrum Badań | |
| Cytat podstawowy | Yates i in. (2020) |
| Dostęp | |
| Strona internetowa | www |
Projekt Ensemble genome database to projekt naukowy Europejskiego Instytutu Bioinformatyki , który został uruchomiony w 1999 roku w odpowiedzi na zbliżające się zakończenie Human Genome Project . Ensembl ma na celu dostarczenie scentralizowanych zasobów dla genetyków, biologów molekularnych i innych badaczy badających genomy naszych własnych gatunków oraz innych kręgowców i organizmów modelowych . Ensembl jest jedną z kilku dobrze znanych przeglądarek genomowych służących do wyszukiwania informacji genomowych .
Podobne bazy danych i przeglądarki można znaleźć w NCBI oraz na Uniwersytecie Kalifornijskim w Santa Cruz (UCSC) .
Tło
Genom ludzki składa się z trzech miliardów par zasad , które kodują około 20 000–25 000 genów . Jednak sam genom jest mało przydatny, chyba że można zidentyfikować lokalizacje i relacje poszczególnych genów. Jedną z opcji jest ręczne dodawanie adnotacji , w ramach którego zespół naukowców próbuje zlokalizować geny, korzystając z danych eksperymentalnych z czasopism naukowych i publicznych baz danych. Jest to jednak powolne, żmudne zadanie. Alternatywą, znany jako zautomatyzowanego adnotacją, jest wykorzystanie mocy obliczeniowych komputerów zrobić kompleksowy wzór dopasowywania z białka do DNA .
W projekcie Ensembl dane sekwencyjne są wprowadzane do systemu adnotacji genów (zbiór „potoków” oprogramowania napisanych w Perlu ), który tworzy zestaw przewidywanych lokalizacji genów i zapisuje je w bazie danych MySQL w celu późniejszej analizy i wyświetlenia. Ensemble udostępnia te dane ogólnoświatowej społeczności naukowej. Wszystkie dane i kody stworzone w ramach projektu Ensembl są dostępne do pobrania, a także dostępny jest publicznie dostępny serwer bazy danych umożliwiający zdalny dostęp. Ponadto witryna internetowa Ensemble zapewnia generowane komputerowo wizualne prezentacje większości danych.
Z biegiem czasu projekt rozszerzył się o dodatkowe gatunki (w tym kluczowe organizmy modelowe, takie jak mysz , muszka owocowa i danio pręgowany ), a także szerszy zakres danych genomowych, w tym wariacje genetyczne i cechy regulacyjne. Od kwietnia 2009 r. siostrzany projekt Ensembl Genomes rozszerzył zakres Ensembl na bezkręgowce metazoa , rośliny , grzyby , bakterie i protisty , podczas gdy pierwotny projekt nadal koncentruje się na kręgowcach.
Wyświetlanie danych genomowych
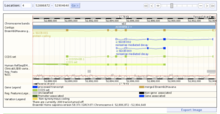
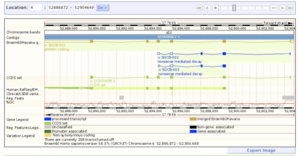
Kluczowym elementem koncepcji Ensembl jest możliwość automatycznego generowania graficznych widoków dopasowania genów i innych danych genomowych względem genomu referencyjnego . Są one wyświetlane jako ścieżki danych, a poszczególne ścieżki można włączać i wyłączać, co pozwala użytkownikowi dostosować wyświetlacz do swoich zainteresowań badawczych. Interfejs umożliwia również użytkownikowi powiększanie regionu lub poruszanie się wzdłuż genomu w dowolnym kierunku.
Inne wyświetlacze pokazują dane na różnych poziomach rozdzielczości, od całych kariotypów po tekstowe reprezentacje sekwencji DNA i aminokwasów , lub prezentują inne rodzaje wyświetlaczy, takie jak drzewa podobnych genów ( homologi ) w różnych gatunkach. Grafiki uzupełniają wyświetlacze tabelaryczne, a w wielu przypadkach dane można wyeksportować bezpośrednio ze strony w różnych standardowych formatach plików, takich jak FASTA .
Dane wytworzone zewnętrznie można również dodać do wyświetlacza, przesyłając odpowiedni plik w jednym z obsługiwanych formatów, takich jak BAM , BED lub PSL .
Grafika jest generowana przy użyciu zestawu niestandardowych modułów Perla opartych na GD , standardowej bibliotece wyświetlania grafiki Perla.
Alternatywne metody dostępu
Oprócz swojej strony internetowej Ensembl zapewnia interfejs API REST i interfejs Perl API ( interfejs programowania aplikacji), który modeluje obiekty biologiczne, takie jak geny i białka, umożliwiając pisanie prostych skryptów w celu pobierania interesujących danych. Ten sam interfejs API jest używany wewnętrznie przez interfejs sieciowy do wyświetlania danych. Jest podzielony na sekcje, takie jak API podstawowe, API porównawcze (dla porównawczych danych genomicznych), API zmienności (dla dostępu do SNP, SNV, CNV...) oraz API genomiki funkcjonalnej (aby uzyskać dostęp do danych regulacyjnych). Witryna Ensemble zawiera obszerne informacje na temat instalacji i korzystania z interfejsu API .
To oprogramowanie może być używane do uzyskiwania dostępu do publicznej bazy danych MySQL , unikając konieczności pobierania ogromnych zestawów danych. Użytkownicy mogą nawet wybrać pobieranie danych z MySQL za pomocą bezpośrednich zapytań SQL, ale wymaga to rozległej wiedzy na temat aktualnego schematu bazy danych.
Duże zbiory danych można pobrać za pomocą narzędzia do eksploracji danych BioMart . Zapewnia interfejs sieciowy do pobierania zestawów danych przy użyciu złożonych zapytań.
Na koniec jest serwer FTP , który może być wykorzystany do pobrania całych baz danych MySQL, a także wybranych zestawów danych w innych formatach.
Aktualne gatunki
Opisane genomy obejmują najbardziej zsekwencjonowane kręgowce i wybrane organizmy modelowe. Wszyscy są eukariontami, nie ma prokariontów. Od 2008 roku obejmuje to:
-
Chordata
-
Mammalia
-
Euarchontoglires
- Naczelne : krzak , szympans , człowiek, makak , lemur myszy , orangutan , wyrak ;
- Scandentia : ryjówka drzewiasta ;
- Gliry (= gryzonie + zajęczaki): świnka morska , szczur kangur , mysz , szczur , wiewiórka , pika , królik ;
- Laurasiatheria : krowa , delfin , alpaka , świnia , kot , pies , koń , megabat , microbat , jeż , ryjówka ;
- Afrotery: słoń , hyrax , Tenrec
- Xenarthra : pancernik , leniwiec ;
- Marsupialia : opos , wallaby ;
- Stekowce : dziobak ;
-
Euarchontoglires
- Ptaki : kurczak , zeberka ;
- Lepidosauria : jaszczurka anolisowa (pre);
- Lissamphibia : Xenopus tropicalis ;
- Teleost rybek Takifugu rubripes ( Fugu ) tetraodon nigroviridis (zielony cętkowany Pufferfish) Danio rerio ( danio pręgowany ) Oryzias latipes ( medaka ) Gasterosteus aculeatus ( ciernik );
- Cyclostomata : Petromyzon marinus ( minóg morski ) (pre);
- osłonice : Ciona intestinalis , Ciona savignyi ;
-
Mammalia
- Bezkręgowce
- Owady : Drosophila melanogaster (mucha owocowa ), Anopheles gambiae (komar), Aedes aegypti (komar)
- Robak : Caenorhabditis elegans
- Drożdże : Saccharomyces cerevisiae (drożdże piekarskie)
Zobacz też
- Lista zsekwencjonowanych genomów eukariotycznych
- Analiza sekwencji
- Narzędzie do profilowania sekwencji
- Motyw sekwencji
- Przeglądarka genomu UCSC
