Mapowanie QTL oparte na rodzinie - Family-based QTL mapping
Ilościowe mapowanie loci cech lub mapowanie QTL to proces identyfikacji regionów genomowych, które potencjalnie zawierają geny odpowiedzialne za ważne cechy ekonomiczne, zdrowotne lub środowiskowe. Mapowanie QTL jest ważną czynnością, którą hodowcy roślin i genetycy rutynowo wykorzystują do powiązania potencjalnych genów przyczynowych z interesującymi fenotypami. Mapowanie QTL oparte na rodzinie jest wariantem mapowania QTL, w którym używa się wielu rodzin.
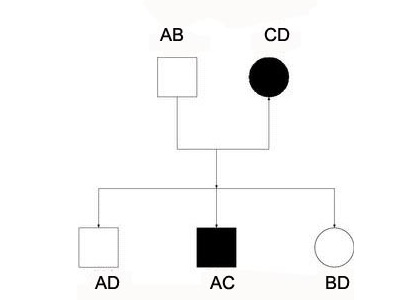
Rodowód u ludzi i pszenicy
Rodowód informacje zawierają informacje o przodkach. Prowadzenie ewidencji rodowodowej to wielowiekowa tradycja. Rodowody można również zweryfikować za pomocą danych markerów genowych.
W roślinach
Metoda została omówiona w kontekście populacji hodowlanych roślin . Rejestry rodowodowe są prowadzone przez hodowców roślin, a selekcja oparta na rodowodach jest popularna w przypadku kilku gatunków roślin. Rodowody roślin różnią się od ludzkich, zwłaszcza że rośliny są hermafrodytyczne – osobnikiem może być samiec lub samica, a kojarzenie może odbywać się w przypadkowych kombinacjach, z pętlami chowu wsobnego. Również rodowody roślin mogą zawierać „ja”, czyli potomstwo powstałe w wyniku samozapylenia rośliny.
Oznaczenie rodowodowe
SIMPLE CROSS SYMBOL Example
/ first order cross SON 64/KLRE
//, second order cross IR 64/KLRE // CIAN0
/3/, third order cross TOBS /3/ SON 64/KLRE // CIAN0
/4/, fourth order cross TOBS /3/ SON 64/KLRE // CIAN0 /4/ SEE
/n/, nth order cross
BACK CROSS SYMBOL
*n n number of times the back cross parent used
left side simple cross symbol,
back cross parent is the female,
right side – male,
Example: SEE/3*ANE, TOBS*6/CIAN0
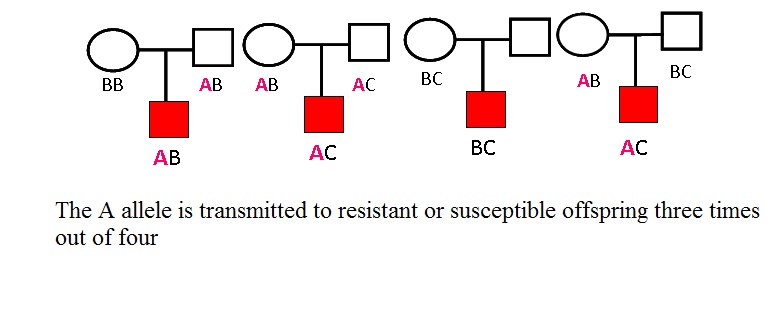
Idea mapowania QTL opartego na rodzinie wywodzi się z dziedziczenia alleli markerowych, a jego powiązanie z interesującą cechą pokazało, jak wykorzystać rodzinne powiązanie w rodzinach hodowlanych roślin.
Ograniczenie metod konwencjonalnych
Tradycyjne populacje mapujące obejmują pojedynczą rodzinę składającą się z krzyżowania między dwojgiem rodziców lub trojgiem rodziców, często daleko spokrewnionych. Istnieje kilka ważnych ograniczeń związanych z tradycyjnymi metodami mapowania. Niektóre z nich obejmują ograniczone wskaźniki polimorfizmu i brak oznak skuteczności markera w wielu podłożach genetycznych. Często, zanim populacja mapująca QTL zostanie opracowana i zmapowana, hodowcy wprowadzają nowy QTL przy użyciu tradycyjnych metod hodowli i selekcji. Może to zmniejszyć użyteczność MAS (selekcja wspomagana markerami) w programach hodowlanych w czasie, gdy MAS może być najbardziej użyteczny (tj. wkrótce po zidentyfikowaniu nowego QTL). Mapowanie QTL oparte na rodzinie usuwa to ograniczenie, wykorzystując istniejące rodziny hodowli roślin.
Wspólne mapowanie populacji badania
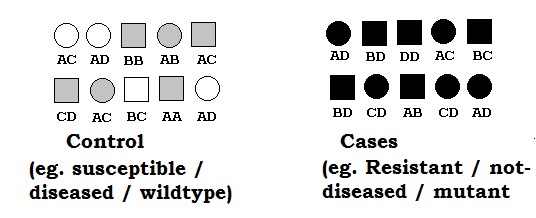
Ogólnie rzecz biorąc, istnieją 3 klasy projektów badań: projekty badań, w których dobierane są próby dużych grup krewnych z rodzin rozszerzonych lub nuklearnych, projekty badań, w których dobierane są próby par krewnych (np. pary rodzeństwa) lub projekty badań, w których dobierane są próby osób niespokrewnionych .
Populacje mapujące stanowi zbiór naturalny osobników (uważanych za niespokrewnione) o nieznanym rodowodzie. Technika mapowania asocjacji w oparciu o populację opiera się na tego typu populacjach. W kontekście roślinnym takie populacje są trudne do znalezienia, ponieważ większość osobników jest w jakiś sposób spokrewniona. Inną wadą takiej metody jest to, że nawet jeśli uda nam się znaleźć taką populację, trudno w takiej sytuacji znaleźć wysoką częstotliwość alleli dla interesującego allelu (zazwyczaj zmutowanego). W celu stworzenia równowagi w częstości alleli, zwykle badania kliniczno-kontrolne.
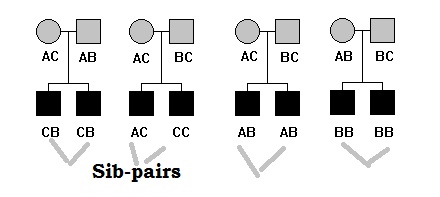
Pary rodzeństwa
Taki projekt obejmuje parę rodzeństwa z wielu niezależnych rodzin. Członkowie każdego rodzeństwa nie są wybierani losowo – często oboje rodzeństwa wybiera się z jednego ogona (górnego lub dolnego) rozkładu QT (rodzeństwo zgodne) lub jedno rodzeństwo jest wybierane z ogona górnego, a drugie rodzeństwo jest wybierane z dolny ogon (niezgodne rodzeństwo). Inny schemat doboru próby może obejmować parę rodzeństwa, jedną wybraną z górnego lub dolnego ogona rozkładu, a drugą wybraną losowo spośród pozostałych rodzeństwa.
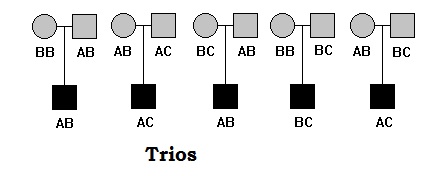
Trio
Trójki obejmują rodziców i jedno potomstwo (najbardziej dotknięte). Tria są częściej używane w badaniach asocjacyjnych. Koncepcja mapowania skojarzeń polega na tym, że każde trio nie jest ze sobą powiązane, jednak tria są ze sobą powiązane.
Rodzina nuklearna
Rodzina nuklearna składa się z dwupokoleniowego rodowodu prostego.
Rozszerzone rodowody
W rozszerzonym rodowodzie zawiera rodowody wielopokoleniowe. Może być tak głęboki lub szeroki, jak dostępne są informacje o rodowodach. Rozszerzony rodowód jest atrakcyjny dla analizy opartej na powiązaniach .
Analiza powiązań a analiza powiązań
Analiza powiązań i asocjacji to podstawowe narzędzia do odkrywania, lokalizacji i analizy funkcjonalnej genów. Chociaż koncepcyjne podstawy tych podejść są znane od dawna, postępy w ostatnich dziesięcioleciach w genetyce molekularnej , rozwój wydajnych algorytmów i moc obliczeniowa umożliwiły zastosowanie tych metod na dużą skalę. Podczas gdy badania powiązań mają na celu identyfikację loci kosegregacji z cechą w rodzinach, badania asocjacyjne mają na celu zidentyfikowanie konkretnych wariantów, które są związane z fenotypem na poziomie populacji. Są to komplementarne metody, które razem zapewniają środki do badania genomu i opisu etiologii złożonych cech . W badaniach powiązań staramy się zidentyfikować loci, które kosegregują z określonym regionem genomowym, oznaczonym markerami polimorficznymi, w obrębie rodzin. Natomiast w badaniach asocjacyjnych poszukujemy korelacji między określoną zmiennością genetyczną a zmiennością cech w próbie osobników, co implikuje przyczynową rolę wariantu.
Analiza powiązań rodzinnych
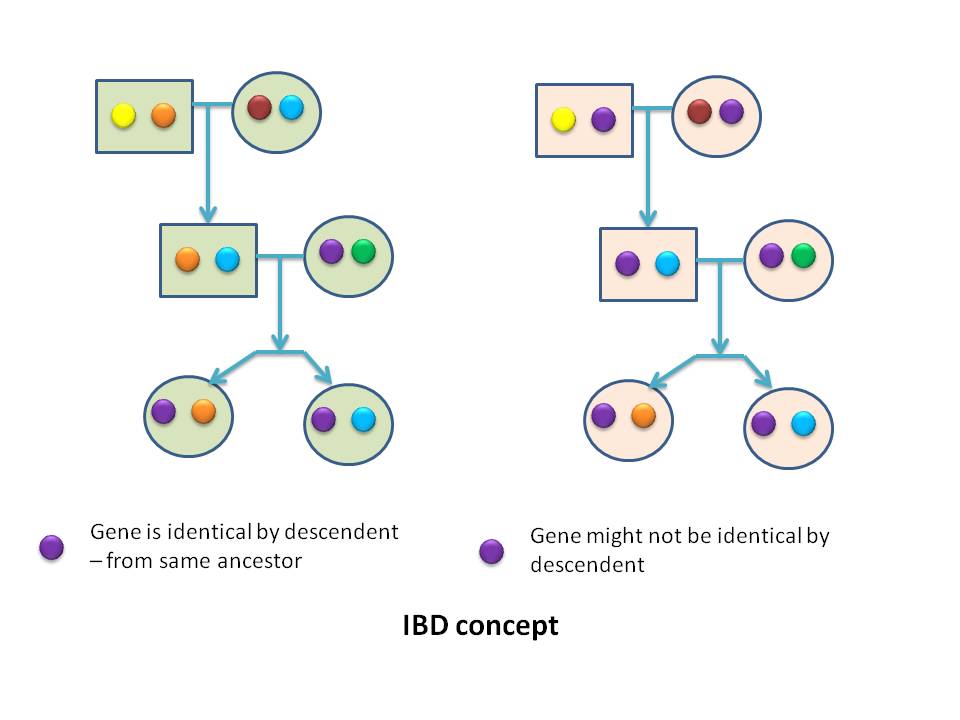
Sprzężenie genetyczne to zjawisko polegające na kosegregacji alleli w różnych loci w rodzinach. Siła kosegregacji jest mierzona frakcją rekombinacji θ, prawdopodobieństwem nieparzystej liczby rekombinacji. Bardziej złożony rodowód zapewnia wyższą moc. Estymacja macierzy tożsamości na podstawie pochodzenia (IBD) jest głównym elementem mapowania loci cech ilościowych (QTL) przy użyciu modeli komponentów wariancji. Allele mają identyczność według typu (IBT), gdy mają ten sam efekt fenotypowy. Allele, które są identyczne pod względem typu, dzielą się na dwie grupy; te, które są identyczne ze względu na pochodzenie (IBD), ponieważ powstały z tego samego allelu we wcześniejszym pokoleniu; oraz te, które nie są identyczne ze względu na pochodzenie (NIBD) lub identyczne ze względu na stan (IBS), ponieważ powstały z oddzielnych mutacji. Pary rodzic-potomstwo mają 50% wspólnych genów IBD, a bliźnięta jednojajowe mają 100% IBD. W analizie sprzężeń istotne jest dziedziczenie (lub współdziedziczenie) alleli w sąsiednich loci; dlatego; krytyczne znaczenie ma ustalenie, czy allele są identyczne pod względem pochodzenia (tj. kopie z tych samych alleli rodzicielskich) czy tylko identyczne pod względem stanu (tj. wyglądają tak samo, ale pochodzą z dwóch różnych kopii alleli). W związku z tym istnieją trzy kategorie analizy powiązań rodzinnych – silnie modelowana (tradycyjny model lod score), słabo oparta na modelu (metody składowych wariancji) lub bez modelu. Metody składowych wariancji mogą być postrzegane jako hybrydy.
Analiza powiązań rodzinnych
Nierównowaga sprzężeń (LD) i mapowanie asocjacyjne cieszą się dużym zainteresowaniem społeczności genetyki roślin ze względu na jej potencjał wykorzystania istniejących kolekcji zasobów genetycznych do dokładnego mapowania loci cech ilościowych (QTL), walidacji genów kandydujących i identyfikacji interesujących alleli (Yu i Buckler, 2006). Trzy elementy o szczególnym znaczeniu dla mapowania asocjacji lub interpretacji wyników to:
- analiza struktury populacji na podgrupy,
- jego zastosowanie do kontroli fałszywych skojarzeń i konsekwencji w konkretnym przypadku zróżnicowanej selekcji między podgrupami, oraz
- analiza lokalnej struktury LD na haplotypy i jej konsekwencje dla rozdzielczości i zastosowania mapowania LD (Flint-Garcia et al. 2003).
W przeciwieństwie do skojarzeń populacyjnych, testy skojarzeń rodzinnych stają się coraz bardziej popularne.
W ostatnich latach dużą popularność zyskał rodzinny test nierównowagi Tran (TDT). Metoda ta również koncentruje się na allelach przenoszonych na potomstwo, ale jest sformułowana z uwzględnieniem zarówno sprzężenia, jak i nierównowagi, które leżą u podstaw tego związku. Test wymaga informacji o genotypie trzech osobników, a mianowicie chorego dziecka i obojga biologicznych rodziców; a co najmniej jeden rodzic musi być heterozygotą, aby test miał charakter informacyjny. Proponowana statystyka testowa jest w rzeczywistości statystyką chi-kwadrat McNemara i testuje hipotezę zerową, że przypuszczalny allel związany z chorobą jest przenoszony w 50% przypadków od heterogygotycznych rodziców w porównaniu z alternatywną hipotezą, że allel związany z cechą dodatnią będzie przenoszony częściej . Na TDT nie ma wpływu stratyfikacja i domieszka populacji. Pojęcie rodzinnego testu skojarzeń zostało rozszerzone na cechy ilościowe.
Ilościowy test nierównowagi transmisji (QTDT)
TDT został rozszerzony w kontekście cech ilościowych i rodzin nuklearnych lub rozszerzonych rodowodowych. Test uogólniony pozwala na użycie w testowaniu dowolnego typu rodziny. QTDT został również rozszerzony o mapowanie asocjacji oparte na haplotypach. Haplotypy odnoszą się do kombinacji alleli markerowych, które są zlokalizowane blisko siebie na tym samym chromosomie i które mają tendencję do wspólnego dziedziczenia. Dzięki dostępności twórców SNP o wysokiej gęstości, haplotypy odgrywają ważną rolę w badaniach asocjacyjnych. Po pierwsze – haplotypy mają kluczowe znaczenie dla zrozumienia wzoru LD w całym genomie, co jest niezbędne do badań asocjacyjnych. Właściwie nie ma lepszego sposobu zrozumienia wzorca LD niż poznanie samych haplotypów. Haplotypy mówią nam, w jaki sposób allele są zorganizowane wzdłuż chromosomu i odzwierciedlają wzór dziedziczenia podczas oceny. Po drugie, metody oparte na haplotypach mogą być silniejsze niż te oparte na pojedynczych markerach w badaniach asocjacyjnych mapowania złożonych genów cech.
Rysowanie rodowodów rodzinnych
Istnieje kilka programów do rysowania rodowodów dostępnych dla kontekstu genetyki człowieka, takich jak COPE (współpraca środowiska rysowania rodowodu), CYRILLIC, FTM (twórca drzewa genealogicznego), FTREE, KINDRED, PED (oprogramowanie do rysowania rodowodu), PEDHUNTER, PEDIGRAPH, PEDIGREE/DRAW, PEDIGREE -VISUALIZER, PEDPLOT,PEDRAW/WPEDRAW (Pedigree Drawing/ Window Pedigree Drawing (wersja PEDRAW MS-Window i X-Window)), PROGENY (Progeny Software, LLC) itp. Jednak rysowanie rodowodowe w roślinach wymaga pewnych dodatkowych funkcji, takich jak chów wsobny, samozapylenie, mutacje, poliploidalność itp., które są obsługiwane w Pedimap . Rodowodowa mapa może być wykorzystana do wizualizacji rodowodu wraz z danymi prawdopodobieństwa fenotypowego, genotypowego i ibd w każdym typie rodowodów roślin, zarówno w diploidach, jak i tetraploidach.
Zobacz też
- Hodowla zwierząt
- Stowarzyszenie genetyczne
- Wybór wspomagany markerem
- Markery molekularne
- Zagnieżdżone mapowanie asocjacji
- Hodowla fizjologiczna i molekularna pszenicy
Bibliografia
- Yu J, Buckler ES (2006) "Genetyczne mapowanie asocjacji i organizacja genomu kukurydzy". Curr Opin Biotechnol 17:155–160
- Flint-Garcia S, Thornsberry JM, Buckler ESIV (2003) „Struktura nierównowagi sprzężeń w roślinach”. Annu Rev Plant Biol 54:357–374