Genetyka i archeogenetyka Azji Południowej - Genetics and archaeogenetics of South Asia
Genetyka i archeogenetyka Azji Południowej jest badanie genetyki i archeogenetyka tych grup etnicznych Azji Południowej . Jego celem jest odkrycie historii genetycznej tych grup . Położenie geograficzne Azji Południowej sprawia, że jej bioróżnorodność jest ważna dla badania wczesnego rozprzestrzeniania się anatomicznie współczesnych ludzi w Azji .
Badania oparte na odmianach mitochondrialnego DNA ( mtDNA ) wykazały jedność genetyczną w różnych subpopulacjach Azji Południowej, pokazując, że większość węzłów przodków drzewa filogenetycznego wszystkich typów mtDNA pochodzi z Azji Południowej. Wnioski z badań opartych na zmienności chromosomu Y i zmienności autosomalnego DNA były zróżnicowane.
Najnowsze badania genomu pokazują, że South Azjaci są potomkami rdzennych komponentu Południowej Azji (nazywane Starożytne Przodków Południowej Indianie „AASI”, luźno związane z Andamanese , Papuasami i Rdzenni Australijczycy), a później przyjazd West-Eurasian (europejski / Middle odpowiednio w różnym stopniu związane ze wschodem) i wschodnioeurazjatyckie (związane z Azją Wschodnią).
Badania genomiczne opisały krajobraz genetyczny Azji Południowej jako połączenie egzogennych komponentów zachodnio- i wschodnio-eurazjatyckich, które mieszały się z rdzennymi grupami południowoazjatyckimi, tworząc współczesnych mieszkańców Azji Południowej. Składnik pochodzenia wschodnioazjatyckiego stanowi główne pochodzenie wśród określonych populacji u podnóża Himalajów i północno - wschodnich Indii i jest ogólnie rozmieszczony na całym subkontynencie indyjskim , osiągając szczyt wśród grup austroazjatyckich , a także wśród syngaleskich i bengalskich .
Stwierdzono, że przodek drzewa filogenetycznego wszystkich typów mtDNA ( haplogrupy mitochondrialnego DNA ) typowo występujących w Azji Środkowej, Zachodniej i Europie można znaleźć również w Azji Południowej ze stosunkowo wysokimi częstotliwościami. Szacuje się, że rozbieżność tego wspólnego węzła przodków miała miejsce nieco mniej niż 50 000 lat temu, wspierając wydarzenie „poza Indiami” dla populacji zachodnio-eurazjatyckich, takich jak Europejczycy, we wczesnym paleolicie. W Indiach, głównych liniach matki różne M subclades, a następnie R, i U sublineages. Czasy koalescencji tych mitochondrialnych haplogrup są do tej pory przybliżone do 50 000 BP.
Główne linie ojcowskie Indian, reprezentowane przez chromosomy Y , to haplogrupy R1a1 , R2 , H , L , J2 i O2a . Niektórzy badacze twierdzili, że haplogrupa Y-DNA R1a1 (M17) ma autochtoniczne pochodzenie południowoazjatyckie. Jednak propozycje dotyczące pochodzenia R1a1 ze stepów środkowoazjatyckich/eurazjatyckich są również dość powszechne i poparte kilkoma nowszymi badaniami.
Przegląd
Sugeruje się, że rdzenna populacja Azji Południowej, czasami określana jako „AASI”, wywodzi się z wczesnej populacji południowego rozproszenia , która dała również początek ludom andamańskim , rdzennym mieszkańcom Nowej Gwinei (Papuans) i rdzennym Australijczykom . Rdzenna ludność Azji Południowej został genetycznie odizolowana od innych populacji od około 45000 lat pne . Rdzenne pochodzenie południowoazjatyckie (AASI) u współczesnych mieszkańców Azji Południowej łączy się, w różnym stopniu, z nowszymi składnikami zachodnio- i wschodnioazjatyckimi z okresu neolitu i epoki brązu . Plemienne Paniya ludzie są, obok ludzi Irula i ludzi Soliga , najlepszym proxy dla rdzennej pochodzenia Południowej Azji.
Sugeruje się, że najwcześniejsze pochodzenie zachodnioazjatyckie pojawiło się w okresie neolitu lub już w holocenie , wraz z przybyciem składników związanych z Azją Wschodnią we wczesnym okresie neolitu z grupami austroazjatyckimi i tybetańsko-birmańskimi odpowiednio z Azji Południowo-Wschodniej i Azji Wschodniej .
mtDNA
Najczęstszymi haplogrupami mtDNA w Azji Południowej są M , R i U (gdzie U jest potomkiem R). Argumentując za długoterminowym „konkurencyjnym modelem chromosomu Y”, Stephen Oppenheimer uważa, że jest to wysoce sugestywne, że Indie są źródłem eurazjatyckich haplogrup mtDNA, które nazywa „euroazjatyckimi epokami”. Według Oppenheimera jest wysoce prawdopodobne, że prawie wszystkie ludzkie linie matczyne w Azji Środkowej, na Bliskim Wschodzie i w Europie wywodzą się od zaledwie czterech linii mtDNA, które powstały w Azji Południowej 50 000-100 000 lat temu.
Makrohaplogrupa M
Macrohaplogroup M , który jest uważany jako klaster proto-azjatyckiej liniach matki, stanowi więcej niż 60% w Azji Południowo mtDNA.
Makrohaplotyp M w Indiach obejmuje wiele podgrup, które znacznie różnią się od innych podlinii w Azji Wschodniej, zwłaszcza populacji mongoloidalnych. Głębokie korzenie filogenezy M wyraźnie wskazują na relikt linii południowoazjatyckich w porównaniu z innymi podliniami M (w Azji Wschodniej i gdzie indziej), co sugeruje pochodzenie tych podhaplogrup „in situ” w Azji Południowej, najprawdopodobniej w Indiach. Te głęboko zakorzenione rodowody nie są specyficzne dla języka i są rozprzestrzenione na wszystkie grupy językowe w Indiach.
Praktycznie wszystkie współczesne środkowoazjatyckie linie MtDNA M wydają się należeć raczej do wschodnioazjatyckich ( mongolskich ) niż południowoazjatyckich podtypów haplogrupy M, co wskazuje, że do Indii nie doszło do migracji na dużą skalę z obecnych tureckojęzycznych populacji Azji Środkowej. Brak haplogrupy M u Europejczyków, w porównaniu z jej równie wysoką częstotliwością wśród mieszkańców Azji Południowej, Azji Wschodniej i niektórych populacji Azji Środkowej, kontrastuje z tendencjami zachodnioazjatyckimi w południowoazjatyckich liniach ojcowskich.
Większość istniejących granic mtDNA w Azji Południowej i Południowo-Zachodniej prawdopodobnie została ukształtowana podczas początkowego zasiedlania Eurazji przez anatomicznie współczesnego człowieka.
| Haplogrupa | Ważne klady podrzędne | Populacje |
|---|---|---|
| M2 | M2a, M2b | Na całym kontynencie z niewielką obecnością w Northwest Peaking w Bangladeszu, Andhra Pradesh, przybrzeżnym Tamil Nadu i Sri Lance |
| M3 | M3a | Skoncentrowany w północno-zachodnich Indiach Najwyższy wśród Parsów Bombaju |
| M4 | M4a | Szczyty w Pakistanie, Kaszmirze i Andhra Pradesh |
| M6 | M6a, M6b | Kaszmir i w pobliżu wybrzeży Zatoki Bengalskiej, Sri Lanka |
| M18 | W całej Azji Południowej Szczyt w Radżastanie i Andhra Pradesh |
|
| M25 | Umiarkowanie częste w Kerali i Maharasztrze, ale raczej rzadkie w innych Indiach |
Makrohaplogrupa R
Macrohaplogroup R (bardzo duża i starych podział macrohaplogroup N ) jest szeroko reprezentowane i stanowi dla pozostałych 40% z Azji Południowo mtDNA. Bardzo starym i najważniejszym pododdziałem jest haplogrupa U, która chociaż występuje również w zachodniej Eurazji , ma kilka podkladów specyficznych dla Azji Południowej.
Najważniejsze haplogrupy południowoazjatyckie w obrębie R:
| Haplogrupa | Populacje |
|---|---|
| R2 | Ukazuje się szeroko na całym subkontynencie |
| R5 | szeroko rozpowszechniony w większości Indii. Szczyty w przybrzeżnych południowo-zachodnich Indiach |
| R6 | rozpowszechnione w niskich cenach w całych Indiach. Szczyty wśród Tamilów i Kaszmirczyków |
| W | Znaleziony w stanach północno-zachodnich. Szczyty w Gujarat, Pendżabie i Kaszmirze, gdzie indziej częstotliwość jest niska. |
Haplogrupa U
Haplogrupa U jest podgrupą makrohaplogrupy R. Rozkład haplogrupy U jest lustrzanym odbiciem tego dla haplogrupy M: ta pierwsza nie została dotychczas opisana wśród wschodnich Azjatów, ale jest częsta w populacjach europejskich, a także wśród południowoazjatyckich. Południowoazjatyckie rodowody U różnią się znacznie od tych w Europie, a ich zlanie się ze wspólnym przodkiem również datuje się na około 50 000 lat.
| Haplogrupa | Populacje |
|---|---|
| U2* | ( parahaplogroup ) jest słabo rozpowszechniony, szczególnie w północnej części Azji Południowej.
Występuje również w południowo-zachodniej Arabii. |
| U2a | wykazuje stosunkowo duże zagęszczenie w Pakistanie i północno-zachodnich Indiach, ale także w Karnatace, gdzie osiąga większe zagęszczenie. |
| U2b | ma najwyższą koncentrację w Uttar Pradesh, ale występuje również w wielu innych miejscach, szczególnie w Kerali i Sri Lance.
Występuje również w Omanie. |
| U2c | jest szczególnie ważne w Bangladeszu i Zachodnim Bengalu. |
| U2l | jest być może najważniejszym pod względem liczbowym subkladem U w Azji Południowej, osiągając szczególnie wysokie stężenia (ponad 10%) w Uttar Pradesh, Sri Lance, Sindh i częściach Karnataki. Ma też pewne znaczenie w Omanie. Haplogrupa mtDNA U2i jest nazwana „Zachodnią Euroazjatycką” w Bamshad et al. badanie, ale „Euroazjatycki Wschodniej (głównie specyficzne dla Indii)” w Kivisild et al. badanie. |
| U7 | ta haplogrupa ma znaczącą obecność w Gujarat, Pendżabie i Pakistanie. Możliwa ojczyzna tej haplogrupy obejmuje Gudżarat (najwyższa frekwencja, 12%) i Iran, ponieważ stamtąd jej frekwencja spada stromo zarówno na wschód, jak i na zachód. |
chromosom Y
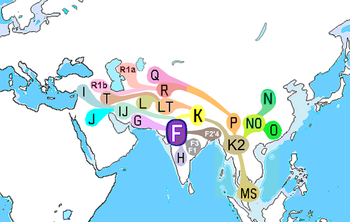
Główne haplogrupy DNA chromosomu Y w Azji Południowej to H , J2 , L , R1a1 , R2 i O-K18 . Ich pochodzenie geograficzne jest wymienione w następujący sposób, zgodnie z najnowszym stypendium:
| Główne południowoazjatyckie linie chromosomu Y: | h | J2 | L | R1a | R2 |
|---|---|---|---|---|---|
| Basu i in. (2003) | bez komentarza | bez komentarza | bez komentarza | Azja centralna | bez komentarza |
| Kivisild i in. (2003) | Indie | Zachodnia Azja | Indie | Azja Południowa i Zachodnia | Azja Południowo-Środkowa |
| Cordaux i in. (2004) | Indie | Azja Zachodnia lub Środkowa | Środkowo Wschodni | Azja centralna | Azja Południowo-Środkowa |
| Sengupta i in. (2006) | Indie | Bliski Wschód i Azja Środkowa | Południowe Indie | Północne Indie | Północne Indie |
| Thanseem i in. (2006) | Indie | Lewant | Bliski wschód | Azja Południowa i Środkowa | Azja Południowa i Środkowa |
| Sahoo i in. (2006) | południowa Azja | Bliski Wschód | południowa Azja | Azja Południowa lub Zachodnia | południowa Azja |
| Mirabal i in. (2009) | bez komentarza | bez komentarza | bez komentarza | Indie Północno-Zachodnie lub Azja Środkowa | bez komentarza |
| Zhao i in. (2009) | Indie | Bliski wschód | Bliski wschód | Azja Środkowa lub Zachodnia Eurazja | Azja Środkowa lub Zachodnia Eurazja |
| Sharma i in. (2009) | bez komentarza | bez komentarza | bez komentarza | południowa Azja | bez komentarza |
| Thangaraj i in. (2010) | południowa Azja | Bliski Wschód | Bliski Wschód | południowa Azja | południowa Azja |
Haplogrupa H
Haplogrupa H (Y-DNA) występuje z dużą częstotliwością w Azji Południowej i jest uważana za reprezentację głównej rodzimej linii ojcowskiej. H jest obecnie rzadko spotykany poza Azją Południową, ale jest powszechny wśród populacji pochodzących z Azji Południowej, takich jak Romowie , szczególnie podgrupa H-M82. H został również znaleziony w niektórych starożytnych próbkach Europy i nadal występuje w niskiej częstotliwości u niektórych południowo-wschodnich Europejczyków i Arabów Lewantu . Haplogrupa H jest często spotykana wśród populacji Indii , Sri Lanki , Nepalu , Pakistanu i Malediwów . Wszystkie trzy gałęzie haplogrupy H (Y-DNA) znajdują się w Azji Południowej.
Uważa się, że haplogrupa H powstała w Azji Południowej między 30 000 a 40 000 lat temu. Jej prawdopodobnym miejscem wprowadzenia jest Azja Południowa, ponieważ tam jest skoncentrowana. Wydaje się, że reprezentuje główną haplogrupę chromosomu Y paleolitycznych mieszkańców odpowiednio Azji Południowej i Europy. Wykazano, że niektóre osobniki w Azji Południowej należą do znacznie rzadszego podkladu H3 (Z5857). Haplogrupa H nie jest w żaden sposób ograniczona do określonych populacji. Na przykład H jest w posiadaniu około 28,8% kast indoaryjskich. aw plemionach około 25-35%.
Haplogrupa J2
Haplogrupa J2 była obecna w Azji Południowej głównie jako J2a-M410 i J2b-M102 od czasów neolitu (9500 YBP). Klady J2 osiągają szczytową częstość występowania w północno-zachodnich i południowych Indiach i występuje w 19% w kastach południowoindyjskich, 11% w kastach północnoindyjskich i 12% w Pakistanie. W południowych Indiach obecność J2 jest wyższa wśród średnich kast (21%), a następnie wyższych (18,6%) i niższych (14%). Wśród grup kastowych najwyższą częstotliwość J2-M172 obserwuje się wśród Tamilów Vellalarów z południowych Indii, na poziomie 38,7%. J2 jest również obecny w plemionach i ma częstotliwość 11% w plemionach austroazjatyckich. Wśród plemion austroazjatyckich dominacja J2 występuje w Lodha (35%). J2 występuje również w południowoindyjskim plemieniu górskim Toda w 38,46%, w plemieniu Andh z Telangana w 35,19% iw plemieniu Kol z Uttar Pradesh z częstotliwością 33,34%. Stwierdzono, że haplogrupa J-P209 jest bardziej powszechna u indyjskich szyickich muzułmanów , z czego 28,7% należy do haplogrupy J, z 13,7% w J-M410, 10,6% w J-M267 i 4,4% w J2b ( Eaaswarkhanth 2009 ) .
W Pakistanie najwyższe częstotliwości J2-M172 pomiędzy zaobserwowano parsowie w 38,89%, w drawidyjskich -speaking Brahuis w 28,18% i Makrani Balochs 24%. Występuje również w 18,18% w Makrani Siddis i 3% w Karnataka Siddis .
J2-M172 występuje w ogólnej częstości 10,3% wśród syngalezi na Sri Lance . Na Malediwach stwierdzono , że 20,6% populacji Malediwów ma haplogrupę J2 dodatnią.
Haplogrupa L
Według dr Spencera Wellsa , L-M20 pochodzi z regionu Pamir Knot w Tadżykistanie i migruje do Pakistanu i Indii ok. 10 tys. 30 000 lat temu. Jednak większość innych badań sugerowała pochodzenie L-M20 z Azji Południowej i wiązała jej ekspansję z doliną Indusu (~7000 YBP). Istnieją trzy podgałęzie haplogrupy L: L1-M76 (L1a1), L2-M317 (L1b) i L3-M357 (L1a2), występujące na różnych poziomach w Azji Południowej.
Indie
Haplogrupa L pokazuje czas ekspansji neolitycznej. Klad występuje w populacji indyjskiej z ogólną częstością ok. 2 tys. 7–15%. Haplogrupa L ma wyższą częstotliwość wśród kast południowoindyjskich (ok. 17–19%) i sięga 68% w niektórych kastach w Karnatace, ale jest nieco rzadsza w kastach północnoindyjskich (ok. 5–6%). Obecność haplogrupy L jest dość rzadka wśród grup plemiennych (ok. 5,6–7%), natomiast wśród czenchów zaobserwowano 14,6% .
Wśród grup regionalnych i społecznych, średnie do wysokich częstotliwości zaobserwowano w konkanastha braminów (18,6%), pendżabskim (12,1%), gudżarati (10,4%), lambadis (17,1%) i dżatach (36,8%).
Pakistan
W Pakistanie podklady L1-M76 i L3-M357 L-M20 osiągają ogólne częstotliwości odpowiednio 5,1% i 6,8%. Haplogrupa L3 (M357) występuje często wśród Buruszy (ok. 12%) i Pasztunów (ok. 7%). Największą częstotliwość można znaleźć w południowo-zachodniej prowincji Beludżystan wzdłuż wybrzeża Makran (28%) do delty rzeki Indus . L3a (PK3) występuje w około 23% Nurystan w północno - zachodnim Pakistanie .
Klad występuje w umiarkowanym rozmieszczeniu wśród ogólnej populacji Pakistanu (ok. 14%).
Sri Lanka
W jednym z badań stwierdzono, że 16% syngaleczyków miało haplogrupę L-M20 dodatnią. W innym badaniu stwierdzono, że 18% należy do L1.
Haplogrupa R1a1
W Azji Południowej R1a1 jest często obserwowany z dużą częstotliwością w wielu grupach demograficznych, a także z największym zróżnicowaniem STR, co skłania niektórych do postrzegania go jako miejsca pochodzenia.
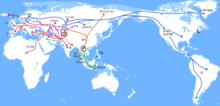
Podczas gdy R1a powstało ca. 22 000 do 25 000 lat temu jego podklad M417 (R1a1a1) zdywersyfikował około. 5800 lat temu. Rozmieszczenie podkladów M417 R1-Z282 (w tym R1-Z280) w Europie Środkowo-Wschodniej i R1-Z93 w Azji sugeruje, że R1a1a zdywersyfikował się w obrębie stepów euroazjatyckich lub regionu Bliskiego Wschodu i Kaukazu . Miejsce pochodzenia tych subkladów odgrywa rolę w debacie o pochodzeniu Indoeuropejczyków . Podczas gdy haplogrupa R jest powszechnie spotykana wśród mieszkańców Europy Zachodniej, ostatnie badania sugerują, że jej rodowa linia ( haplogrupa P ) wywodzi się z populacji południowo-eurazjatyckiej w Azji Południowo-Wschodniej . Dziś jest powszechnie spotykany wśród ludów Aeta , Jahai i niektórych ludów andamańskich .
Indie
W Indiach wysoki procent tej haplogrupy obserwuje się u braminów Zachodniego Bengalu (72%) na wschodzie, Gujarat Lohanas (60%) na zachodzie, Khatris (67%) na północy i Iyengara braminów (31%) w południe. Została również znaleziona w kilku południowoindyjskich plemionach posługujących się językiem drawidyjskim, w tym Kotas (41%) z Tamil Nadu, Chenchu (26%) i Valmikis z Andhra Pradesh, a także Yadav i Kallar z Tamil Nadu, co sugeruje, że M17 jest szeroko rozpowszechniona w tych południowych plemion Indian. Poza tym badania wykazują wysokie odsetki w zróżnicowanych regionalnie grupach, takich jak Manipuris (50%) na skrajnym północnym wschodzie i wśród Pendżabów (47%) na skrajnym północnym zachodzie.
Pakistan
W Pakistanie występuje w 71% w prowincji Mohanna z Sindh na południu i 46% w Baltis z Gilgit-Baltistan na północy.
Sri Lanka
23% syngalezi spośród próbie 87 pacjentów uznano za R1a1a (R-SRY1532) dodatni Według badań 2003 r.
Malediwy
Na Malediwach 23,8% mieszkańców Malediwów miało R1a1a (M17) dodatni.
Nepal
Ludzie w regionie Terai w Nepalu wykazują R1a1a na poziomie 69%.
Haplogrupa R2
W Azji Południowej częstość linii R2 i R2a wynosi około 10–15% w Indiach i Sri Lance oraz 7–8% w Pakistanie. Co najmniej 90% osobników R-M124 znajduje się w Azji Południowej. Z mniejszą częstotliwością odnotowuje się go również na Kaukazie iw Azji Środkowej . Badanie genetyczne przeprowadzone przez Mondal et al. w 2017 roku stwierdził, że Haplogroup R2 pochodzi z północnych Indii i była obecna przed migracją stepową.
Indie
Wśród grup regionalnych znajduje się wśród Bengalczyków Zachodnich (23%), Hindusów z New Delhi (20%), Pendżabów (5%) i Gudżarati (3%). Wśród grup plemiennych Karmali z Zachodniego Bengalu wykazali najwyższy poziom 100%, a następnie Lodhas (43%) na wschodzie, podczas gdy Bhil z Gujarat na zachodzie mieli 18%, Tharus z północy pokazał to 17%, Chenchu i Pallan południa wynosiły odpowiednio 20% i 14%. Wśród grup kastowych wysoki odsetek wykazują Jaunpur Kshatriyas (87%), Kamma (73%), Bihar Yadav (50%), Khandayat (46%) i Kallar (44%).
Jest również znacznie wysoki w wielu grupach bramińskich , w tym braminach pendżabskich (25%), braminach bengalskich (22%), braminach konkanastha (20%), chaturwedi (32%), bhargawach (32%), kaszmirskich panditach (14%) i Lingayat Bramini (30%).
Północnoindyjscy muzułmanie mają częstotliwość 19% ( sunnici ) i 13% ( szyici ), podczas gdy muzułmanie Dawoodi Bohra w zachodnim stanie Gujarat mają częstotliwość 16%, a muzułmanie Mappila z południowych Indii mają częstotliwość 5%.
Pakistan
Haplogrupa R2 znajduje się u 71% mieszkańców Burusho . Wśród ludu Hunza znajduje się on na 18%, podczas gdy Parsowie pokazują go na 20%.
Sri Lanka
Według badań z 2003 r. 38% syngaleskich mieszkańców Sri Lanki miało wynik R2 dodatni.
Malediwy
12% Malediwów na Malediwach ma R2.
Nepal
W Nepalu wartości procentowe R2 wahają się od 2% do 26% w różnych grupach w ramach różnych badań. Newars wykazuje znacząco wysoką częstotliwość 26%, podczas gdy mieszkańcy Katmandu wykazują ją na poziomie 10%.
Haplogrupa O2a (O-K18)
W Azji Południowej średnia częstotliwość występowania O2a lub O-K18 wynosi odpowiednio 5%-15% w Indiach i 15%-35% na Sri Lance i Bangladeszu. Wśród grup regionalnych, zwłaszcza wśród grup austroazjatyckich, osiąga szczyt na poziomie około 75%. Haplogrupa O2a (O-K18) jest często spotykana wśród Azji Południowo-Wschodniej i Wschodniej, zwłaszcza wśród austroazjatyckich rolników zajmujących się uprawą ryżu. Sugeruje się, że O2a (O-K18) przybył z Azji Południowo-Wschodniej do Indii po ostatnim maksimum lodowcowym, około 10 000 p.n.e.
Rekonstrukcja historii populacji Azji Południowej
Indian Genome Variation Consortium (2008) , dzieli mieszkańców Azji Południowej na cztery etnolingwistycznych (nie genetyczne) grupy: indoeuropejskich , drawidyjskich , Tibeto-Burman i Austro-azjatyckiej . Badania antropologii molekularnej wykorzystują trzy różne typy markerów: zmienność mitochondrialnego DNA (mtDNA), która jest dziedziczona po matce i wysoce polimorficzna, zmienność chromosomu Y, która obejmuje transmisję jednorodzicielską wzdłuż linii męskich oraz zmienność autosomalnego DNA.
odmiana mtDNA
Większość badań opartych na zmienności mtDNA wykazała jedność genetyczną populacji południowoazjatyckich w grupach językowych, kastowych i plemiennych. Jest prawdopodobne, że haplogrupa M została przywieziona do Azji z Afryki Wschodniej wzdłuż południowej trasy przez najwcześniejszą falę migracji 78 000 lat temu.
Według Kivisilda i in. (1999), „Niewielkie pokrywanie się z rodowodami opisanymi w innych populacjach euroazjatyckich wyraźnie pokazuje, że niedawne imigracje miały bardzo mały wpływ na wrodzoną strukturę macierzystej puli genowej Azjatów Południowych. Pomimo różnic stwierdzonych w Indiach, populacje te wywodzą się z ograniczonej liczba linii założycieli. Te linie zostały najprawdopodobniej wprowadzone do Azji Południowej w środkowym paleolicie, przed zaludnieniem Europy 48 000 lat temu i być może w ogóle Starego Świata”. Basu i in. (2003) podkreśla również podstawową jedność linii żeńskich w Indiach.
Odmiana chromosomu Y
Wnioski oparte na zmienności chromosomu Y były bardziej zróżnicowane niż te oparte na zmienności mtDNA. Podczas gdy Kivisild i in. (2003) proponuje starożytne i wspólne dziedzictwo genetyczne męskich linii genetycznych w Azji Południowej, Bamshad et al. (2001) sugeruje pokrewieństwo między południowoazjatyckimi męskimi rodami a współczesnymi zachodnimi Euroazjatami proporcjonalne do rangi wyższej kasty i umieszcza populacje wyższych kast w południowych stanach Indii bliżej wschodnioeuropejskich.
Basu i in. (2003) konkluduje, że austro-azjatyckie populacje plemienne przybyły do Indii najpierw z korytarza północno-zachodniego, a znacznie później niektóre z nich przez korytarz północno-wschodni. Natomiast Kumar i in. (2007) przeanalizowali 25 południowoazjatyckich plemion austroazjatyckich i stwierdzili silny związek genetyczny ojcowski wśród grup podjęzykowych południowoazjatyckich populacji austroazjatyckich. Mukherjee i in. (2001) umieszcza Pakistańczyków i Indian północnych między populacjami zachodnioazjatyckimi i środkowoazjatyckimi, podczas gdy Cordaux et al. (2004) twierdzi, że populacje kast indyjskich są bliższe populacji Azji Środkowej. Sahoo i in. (2006) oraz Sengupta i in. (2006) sugerują, że populacje kast indyjskich nie zostały poddane żadnym ostatnim domieszkom. Sanghamitra Sahoo podsumowuje swoje badanie:
Opierając się na aktualnych dowodach, nie jest konieczne szukanie poza Azją Południową pochodzenia ojcowskiego dziedzictwa większości Indian w czasie początków osiadłego rolnictwa. Odwieczna koncepcja ludzi, języka i rolnictwa przybywających razem do Indii korytarzem północno-zachodnim nie wytrzymuje dokładnej analizy. Niedawne twierdzenia o powiązaniu haplogrup J2, L, R1a i R2 ze współczesnym pochodzeniem większości linii ojcowskich kast indyjskich spoza Azji Południowej są odrzucane, chociaż nasze ustalenia potwierdzają lokalne pochodzenie haplogrup F* i H. Spośród pozostałych tylko J2 wskazuje na jednoznaczny niedawny wkład zewnętrzny, z Azji Zachodniej, a nie z Azji Środkowej. Obecne rozkłady częstości haplogrup są, z wyjątkiem linii genealogicznych, głównie determinowane przez uwarunkowania geograficzne, a nie kulturowe. Jak na ironię, to właśnie w północno-wschodnich Indiach, wśród grup gruźlicy, istnieją wyraźne dowody na dyfuzję demiczną na dużą skalę, którą można prześledzić za pomocą genów, kultury i języka, ale najwyraźniej nie przez rolnictwo.
Analiza najbliższego sąsiedztwa przeprowadzona przez Mondal et al. w 2017 roku stwierdził, że indyjski Y rodowody są zbliżone do południowych europejskich społeczeństw oraz czas rozbieżności pomiędzy dwoma wyprzedził migracji Step:
Wyniki te sugerują, że europejskie pochodzenie w populacjach indyjskich może być znacznie starsze i bardziej złożone niż oczekiwano i może pochodzić z pierwszej fali rolników lub nawet wcześniej
— Mondal i in. 2017
Autosomalna zmienność DNA
AASI-ANI-ASI
Zróżnicowane były również wyniki badań opartych na autosomalnej zmienności DNA. W dużym badaniu (2009) wykorzystującym ponad 500 000 biallelicznych markerów autosomalnych, Reich postawił hipotezę, że współczesna populacja Azji Południowej była wynikiem domieszki dwóch genetycznie rozbieżnych populacji przodków datowanych na epokę postholocenową. Te dwie "zrekonstruowane" starożytne populacje nazwał "Przodkami Południowych Indian" (ASI) i "Przodkami Północnych Indian" (ANI). Według Reicha: „Pochodzenie ANI jest znacznie wyższe w językach indoeuropejskich niż drawidyjskich, co sugeruje, że przodkowie ASI mogli mówić językiem drawidyjskim przed zmieszaniem się z ANI”. Podczas gdy ANI jest genetycznie zbliżone do mieszkańców Bliskiego Wschodu, Azji Środkowej i Europejczyków, ASI nie jest blisko spokrewniony z grupami spoza subkontynentu. Ponieważ nie jest dostępne starożytne DNA „ASI”, rdzenni andamańscy Onge są wykorzystywani jako (niedoskonałe) zastępstwo ASI (według Reicha i in., Andamańczycy, choć różnią się od nich, są najbliższą żyjącą populacją ASI). Według Reicha i wsp., zarówno pochodzenie ANI, jak i ASI występuje na całym subkontynencie (zarówno w północnych, jak i południowych Indiach) w różnych proporcjach, a „pochodzenie ANI waha się od 39-71% w Indiach i jest wyższe w tradycyjnie górnych posłowie kastowi i indoeuropejscy."
Moorjani i in. 2013 stwierdza, że ASI, choć nie są blisko spokrewnieni z żadną żyjącą grupą, są „spokrewnieni (daleko) z rdzennymi Andamanami”. Moorjani i in. jednak zasugeruj możliwy przepływ genów do Andamańczyków z populacji spokrewnionej z ASI, powodując modelowany związek. Badanie wykazało, że „prawie wszystkie grupy posługujące się językami indoeuropejskimi lub drawidyjskimi leżą na gradiencie różnego pokrewieństwa z zachodnio-eurazjatami w PCA (określanym jako „klina indyjska”)”.
Badanie przeprowadzone przez Chaubeya z 2013 roku przy użyciu polimorfizmu pojedynczego nukleotydu (SNP) pokazuje, że genom Andamańczyków (Onge) jest bliższy genom innych grup Oceanic Negrito niż genom Azji Południowej.
Według Basu i in. W 2016 r. dalsza analiza wykazała, że strukturę genomową populacji Indii kontynentalnych najlepiej można wytłumaczyć wkładem czterech składników przodków. Oprócz ANI i ASI Basu i in. al (2016) zidentyfikowali dwa wschodnioazjatyckie elementy przodków w Indiach kontynentalnych, które są główne dla plemion mówiących w języku austro-azjatyckim i mówców tybetańsko-birmańskich, które oznaczyli jako AAA (od „przodków austroazjatyckich”) i ATB (od „ przodków Tybeto-Birmana”). Z badania wynika również, że populacje archipelagu Wysp Andamańskich tworzą odrębne pochodzenie, które „okazało się być rodem z populacji oceanicznych ”, ale bardziej odległe od Azji Południowej.
Linia domieszki pomiędzy liniami ANI i ASI datowana jest na okres ok. 4,2–1,9 kya Moorjaniego i in. (2013), odpowiadające indyjskiej epoce brązu i kojarzone przez autorów z procesem dezurbanizacji Cywilizacji Doliny Indusu i przesunięciem populacji do systemu Gangesu w początkowej indyjskiej epoce żelaza. Basu i in. (2003) sugeruje, że „mówcy drawidyjscy byli prawdopodobnie szeroko rozpowszechnieni w Indiach przed przybyciem koczowników mówiących w języku indoeuropejskim” i że „tworzenie populacji przez rozszczepienie, które spowodowało skutki założycielskie i dryfujące, odcisnęło swoje piętno na strukturach genetycznych współczesnych populacje". Genetyk PP Majumder (2010) twierdził niedawno, że odkrycia Reicha i in. (2009) są w niezwykłej zgodności z wcześniejszymi badaniami z wykorzystaniem mtDNA i Y-DNA:
Przypuszcza się, że populacje środkowoazjatyckie miały duży wkład w indyjską pulę genową, szczególnie w północnoindyjską pulę genową, a migranci rzekomo przenieśli się do Indii przez tereny dzisiejszego Afganistanu i Pakistanu. Korzystając z danych dotyczących zmienności mitochondrialnego DNA zebranych z różnych badań, wykazaliśmy, że populacje Azji Środkowej i Pakistanu wykazują najniższy współczynnik zróżnicowania genetycznego z populacjami północnoindyjskimi, wyższe z populacjami południowych Indii, a najwyższy z populacjami północno-wschodnimi Indii . Populacje północnych Indii są genetycznie bliżej Azji Środkowej niż populacje innych regionów geograficznych Indii... . Zgodnie z powyższymi odkryciami, niedawne badanie wykorzystujące ponad 500 000 autosomalnych markerów biallelicznych wykazało gradient bliskości genetycznej populacji indyjskich z zachodnimi Euroazjatami z północy na południe. Ta cecha jest prawdopodobnie związana z proporcjami pochodzenia wywodzącego się z puli genów zachodniej Eurazji, która, jak pokazało to badanie, jest większa w populacjach zamieszkujących Indie północne niż w Indiach południowych.
Chaubey i in. W 2015 r. wykryto charakterystyczny element przodków Azji Wschodniej, ograniczony głównie do określonych populacji u podnóża Himalajów i północno-wschodniej części Indii. Najwyższa częstotliwość tego składnika jest obserwowana wśród tybetańsko-birmańskich grup w północno-wschodnich Indiach i została również wykryta w populacjach andamańskich na poziomie 32%, przy czym znaczna obecność występuje również wśród osób posługujących się językiem austroazjatyckim. Stwierdzono, że jest on w dużej mierze nieobecny w głośnikach indoeuropejskich i drawidyjskich, z wyjątkiem niektórych określonych grup etnicznych żyjących u podnóża Himalajów i środkowo-południowych Indii. Badacze zasugerowali jednak, że pochodzenie wschodnioazjatyckie (reprezentowane przez Han) mierzone w badanych grupach andamańskich może w rzeczywistości odzwierciedlać uchwycenie powinowactwa Andamańczyków z Melanezyjczykami i malezyjskimi Negrito (zamiast prawdziwej domieszki wschodnioazjatyckiej), jak w poprzednim badaniu przez Chaubey et al. zasugerował „głębokie wspólne pochodzenie” między Andamańczykami, Melanezyjczykami i innymi grupami Negrito oraz powinowactwo między południowo-wschodnioazjatyckimi Negrito i Melanezyjczykami (a także Andamańczykami) z Azjatami z Azji Wschodniej.
Lazaridis i in. (2016) zauważa: „Wpływ demograficzny populacji związanych ze stepem na Azję Południową był znaczny, ponieważ Mala , południowoindyjska populacja Dalitów z minimalnym pochodzeniem z północnych Indii (ANI) nadal ma ~ 18% pochodzenia Yamnaya związanego ze stepem , podczas gdy Kalash Sugeruje się, że Pakistan ma około 50% pochodzenia Yamnaya związanego ze stepem, co stanowi jeden z najwyższych odsetków obok mieszkańców Europy Wschodniej (51%). W badaniu Lazaridis i wsp. z 2016 r. oszacowano (6,5–50,2%) domieszkę związaną ze stepem u mieszkańców Azji Południowej. Lazaridis i in. zauważa ponadto, że „przydatnym kierunkiem przyszłych badań jest bardziej kompleksowe pobieranie próbek starożytnego DNA z populacji stepowych, a także populacji Azji Środkowej (na wschód od Iranu i na południe od stepu), które może ujawnić bardziej bezpośrednie źródła ANI niż tych rozważanych tutaj, a także Azji Południowej, aby bezpośrednio określić trajektorię zmian demograficznych na tym obszarze.
Pathak i in. W 2018 r. stwierdzono, że osoby posługujące się językiem indoeuropejskim z równin Gangesu i osoby posługujące się drawidyjczykami mają znaczny rodowód Yamnaya wczesno-środkowej epoki brązu (Steppe_EMBA), ale nie mają przodków w środkowej i późnej epoce brązu (Steppe_MLBA). Z drugiej strony populacje „północno-zachodnich Indii i Pakistanu” (PNWI) wykazywały znaczące pochodzenie Steppe_MLBA wraz z pochodzeniem Yamnaya (Steppe_EMBA). Badanie wykazało również, że starożytne próbki z Azji Południowej miały znacznie wyższą wartość Steppe_MLBA niż Steppe_EMBA (lub Yamnaya). Badanie sugerowało również, że Rors może być używany jako zastępstwo dla ANI.
David Reich w swojej książce z 2018 r. Kim jesteśmy i jak się tutaj znajdujemy stwierdza, że analizy z 2016 r. wykazały, że ASI ma znaczne ilości składnika pochodzenia pochodzącego od irańskich rolników (około 25% ich przodków), a pozostałe 75% ich przodków pochodzenie wywodzące się od rodzimych południowoazjatyckich łowców-zbieraczy. Dodaje, że ASI były mało prawdopodobne jako lokalni łowcy-zbieracze w Azji Południowej, jak wcześniej ustalono, ale populacja odpowiedzialna za rozprzestrzenianie rolnictwa w całej Azji Południowej. W przypadku ANI pochodzenie irańskich rolników wynosi 50%, a reszta pochodzi z grup stepowych związanych z Yamnaya.
Narasimhan i in. (2018) , podobnie konkludują, że ANI i ASI powstały w II tysiącleciu p.n.e. Poprzedziła ich mieszanka AASI (starożytni przodkowie południowych Indii, tj. myśliwi-zbieracze mający odległe korzenie z Andamańczykami, australijskimi Aborygenami i Azjatami Wschodnimi); i irańscy rolnicy, którzy przybyli do Indii około. 4700-3000 p.n.e. i „musi dotrzeć do Doliny Indusu w 4 tysiącleciu pne”. Według Narasimhana i in. ta mieszana populacja, prawdopodobnie pochodząca z cywilizacji doliny Indusu, „przyczyniła się w dużym stopniu zarówno do ANI, jak i ASI”, która ukształtowała się w drugim tysiącleciu p.n.e. ANI powstało z mieszaniny „ grup związanych z peryferiami Indusu ” i migrantów ze stepów, podczas gdy ASI powstało z „ grup związanych z peryferiami Indusu ”, które przeniosły się na południe i mieszały dalej z lokalnymi zbieraczami-łowcami. Sugeruje się, że pochodzenie populacji ASI wynosiło średnio około 73% od AASI i 27% od rolników związanych z Iranem. Narasimhan i in. zauważyć, że próbki z peryferii Indusu są zawsze mieszanką tych samych dwóch bliższych źródeł pochodzenia AASI i irańskich rolników; z „jedną z osób z Indus Periphery mającą ~ 42% pochodzenia AASI, a pozostałe dwie osoby mające ~ 14-18% pochodzenia AASI” (przy czym pozostała część ich przodków pochodzi z irańskiej populacji związanej z rolnikami). Autorzy sugerują, że rdzenni łowcy-zbieracze AASI reprezentują rozbieżną gałąź, która oddzieliła się mniej więcej w tym samym czasie, w którym oddzielili się od siebie przodkowie z Azji Wschodniej, Onge (Andamańczycy) i australijscy Aborygeni. Wywnioskował, że „zasadniczo całe pochodzenie dzisiejszych wschodnich i południowych Azjatów (przed domieszką związaną z zachodnią Europą u południowych Azjatów) wywodzi się z pojedynczego rozprzestrzeniania się na wschód, co dało początek w krótkim czasie rodowodom prowadzącym do AASI , Azji Wschodniej, Onge i Australijczyków”.
Badanie genetyczne przeprowadzone przez Yelmena i in. (2019) pokazuje jednak, że współczesne populacje południowoazjatyckie są na ogół najbliżej siebie i dość różnią się od populacji poza Azją Południową. Doszli do wniosku, że współcześni mieszkańcy Azji Południowej są w zasadzie mieszanką rodzimego komponentu południowoazjatyckiego (AASI) i przybywającego później komponentu zachodnioazjatyckiego (pochodzącego zarówno z Azji Zachodniej, jak i zachodnich Stepów). Autorzy ujawnili również, że rdzenny komponent południowoazjatycki (AASI) różni się raczej od Andamańczyków lub mieszkańców Azji Wschodniej, a zatem Andamańczycy są niedoskonałym odpowiednikiem. Proponują, że południowoindyjskie plemię Paniya (grupa pochodzenia w przeważającej części z ASI) służyłoby jako lepsze zastępstwo niż Andamańczycy (Onge) dla „rdzennego południowoazjatyckiego” komponentu współczesnych Azjatów Południowych, ponieważ Paniya wywodzą się bezpośrednio z tubylcy z Azji Południowej, podczas gdy Onge może otrzymać przepływ genów od innych grup. Odkryli, że rdzenna populacja Azji Południowej była izolowana od innych populacji od co najmniej 45 000 pne.
Dwa badania genetyczne (Shinde i in. 2019 oraz Narasimhan i in. 2019) analizujące szczątki z cywilizacji Doliny Indusu (części północno-zachodnich Indii i Wschodniego Pakistanu z epoki brązu) wykazały, że mają one mieszankę przodków, zarówno z rodzimego południa Azjatyccy myśliwi-zbieracze dzielący odległe korzenie z Andamańczykami oraz z grupy spokrewnionej z irańskimi rolnikami. Próbki przeanalizowane przez Shinde pochodziły około 50-98% ich genomu z ludów spokrewnionych z Iranem i od 2-50% od rdzennych południowoazjatyckich łowców-zbieraczy. Próbki analizowane przez Narasimhana i in. miał 45-82% irańskiego pochodzenia rolników i 11-50% pochodzenia południowoazjatyckiego myśliwego-zbieracza. Analizowane próbki z obu badań nie mają prawie żadnego składnika „ pochodzenia stepowego ” związanego z późniejszymi migracjami indoeuropejskimi do Indii. Autorzy stwierdzili, że odpowiednie ilości tych przodków różniły się znacznie między osobnikami i doszli do wniosku, że potrzeba więcej próbek, aby uzyskać pełny obraz historii populacji indyjskiej.
Dystans genetyczny między grupami kastowymi a plemionami
Badania Watkinsa i in. (2005) oraz Kivisild i in. (2003) na podstawie markerów autosomalnych doszli do wniosku, że populacje kastowe i plemienne mają wspólnego przodka. Reddy i in. (2005) stwierdzili dość równomierne rozkłady częstotliwości alleli w grupach kastowych w południowym Andhra Pradesh , ale znacznie większy dystans genetyczny między grupami kastowymi i plemionami, co wskazuje na izolację genetyczną plemion i kast.
Viswanathan i in. (2004) w badaniu struktury genetycznej i powinowactwa między populacjami plemiennymi w południowych Indiach konkluduje: „ Zróżnicowanie genetyczne było wysokie, a odległości genetyczne nie były istotnie skorelowane z odległościami geograficznymi. Dlatego dryf genetyczny prawdopodobnie odegrał znaczącą rolę w kształtowaniu wzorców zmienności genetycznej zaobserwowano w populacjach plemion południowoindyjskich. Poza tym analizy relacji populacyjnych wykazały, że wszystkie populacje indyjskie i południowoazjatyckie są nadal do siebie podobne, niezależnie od cech fenotypowych i nie wykazują żadnego szczególnego podobieństwa do Afrykanów. Wnioskujemy, że podobieństwa fenotypowe niektóre grupy indyjskie na rzecz Afrykanów nie odzwierciedlają bliskiego związku między tymi grupami, ale można je lepiej wyjaśnić konwergencją ”.
Badanie z 2011 roku opublikowane w American Journal of Human Genetics wskazuje, że indyjskie elementy przodków są wynikiem bardziej złożonej historii demograficznej niż wcześniej sądzono. Według naukowców Azja Południowa posiada dwa główne komponenty przodków, z których jeden rozprzestrzenia się z porównywalną częstotliwością i różnorodnością genetyczną w populacjach Azji Środkowej, Zachodniej i Europy; drugi składnik jest bardziej ograniczony do Azji Południowej. Gdyby jednak wykluczyć możliwość wielkoskalowej migracji indoaryjskiej, odkrycia te sugerują, że powinowactwo genetyczne obu indyjskich składników przodków jest wynikiem wielu przepływów genów na przestrzeni tysięcy lat.
Modelowanie obserwowanych zróżnicowań haplotypów sugeruje, że oba komponenty indyjskiego pochodzenia są starsze niż rzekoma inwazja indoaryjska 3500 YBP. Zgodnie z wynikami parzystych odległości genetycznych między regionami świata, Indianie mają więcej sygnałów przodków z Zachodem niż z wschodnioeurazjatami.
Narashimhan i in. 2019 stwierdzili, że austroazjatyckich plemion Munda nie można modelować po prostu jako mieszankę przodków ASI, AASI, ANI, w przeciwieństwie do innych mieszkańców Azji Południowej, ale wymagały dodatkowego elementu przodków z Azji Południowo-Wschodniej. Zostały one wymodelowane jako mieszanina 88% AASI i 12% wschodnioazjatyckich spokrewnionych z Nicobarese , zatem profil przodków Mundas zapewnia niezależną linię przodków z Azji Południowo-Wschodniej około trzeciego tysiąclecia pne. Lipson i in. W 2018 r. znaleziono podobne wyniki domieszek w odniesieniu do plemion Munda, stwierdzając, że „uzyskaliśmy dobre dopasowanie z trzema składnikami przodków: jednym zachodnio-euroazjatyckim, jednym głęboko wschodnim euroazjatyckim (interpretowanym jako rdzenny rodowód południowoazjatycki) i jednym z kladu austroazjatyckiego” . Lipson i in. W 2018 r. stwierdzono ponadto, że austroazjatyckie źródło odziane (proporcja 35%) w plemionach Munda zostało uznane za najbliższe Mlabri . Singh i wsp. 2020 podobnie stwierdzili, że austroazjatyckie osoby mówiące w Azji Południowej wypadają z linii południowoazjatyckiej ze względu na ich powinowactwo genetyczne do Azji Południowo-Wschodniej.
Zobacz też
- Archeogenetyka
- Grupy etniczne Azji Południowej
- Lista regionów etnolingwistycznych Azji Południowej
- Ludność Indii
- Haplogrupy Y-DNA w populacjach Azji Południowej
- Badania genetyczne na ludziach Gujarati
- Genetyczna historia Europy
- Genetyczna historia Bliskiego Wschodu
Uwagi
Bibliografia
Dalsza lektura
- Allikas A, Ord D, Kurg R, Kivi S, Ustav M (czerwiec 2001). „Role regionu zawiasowego i domeny wiążącej DNA białka E2 wirusa brodawczaka bydła typu 1 w inicjacji replikacji DNA”. Badania wirusów . 75 (2): 95-106. doi : 10.1016/S0168-1702(01)00219-2 . PMID 11325464 .
- Behar DM, Garrigan D, Kaplan ME, Mobasher Z, Rosengarten D, Karafet TM i in. (marzec 2004). „Kontrastowe wzory zmienności chromosomu Y w aszkenazyjskich żydowskich i gospodarzach nieżydowskich populacji europejskich”. Genetyka człowieka . 114 (4): 354–65. doi : 10.1007/s00439-003-1073-7 . PMID 14740294 . S2CID 10310338 .
- Bhattacharyya NP, Basu P, Das M, Pramanik S, Banerjee R, Roy B, et al. (sierpień 1999). „Znikomy przepływ męskich genów przez granice etniczne w Indiach, ujawniony przez analizę polimorfizmów chromosomu Y DNA” . Badania genomu . 9 (8): 711–9. doi : 10.1101/gr.9.8.711 (nieaktywny 6 maja 2021 r.). PMID 10447506 .CS1 maint: DOI nieaktywny od maja 2021 ( link )
- Cann RL (marzec 2001). „Genetyczne wskazówki dotyczące rozproszenia w populacjach ludzkich: odtworzenie przeszłości z teraźniejszości”. Nauka . 291 (5509): 1742-8. Kod Bibcode : 2001Sci...291.1742C . doi : 10.1126/science.1058948 . PMID 11249820 . S2CID 19367408 .
- Cinnioğlu C, King R, Kivisild T, Kalfoğlu E, Atasoy S, Cavalleri GL, et al. (styczeń 2004). „Wykopywanie warstw haplotypów chromosomu Y w Anatolii”. Genetyka człowieka . 114 (2): 127–48. doi : 10.1007/s00439-003-1031-4 . PMID 14586639 . S2CID 10763736 .
- Das B, Chauhan PS, Seshadri M (październik 2004). „Minimalne współdzielenie haplotypów STR chromosomu Y wśród pięciu endogamicznych grup ludności z zachodnich i południowo-zachodnich Indii”. Biologia człowieka . 76 (5): 743–63. doi : 10.1353/hub.2005.003 . PMID 15757245 . S2CID 12835244 .
- Hemphill BE, Christensen AF (3 listopada 1994). Cywilizacja Oxus jako łącznik między Wschodem a Zachodem: niemetryczna analiza powinowactwa biologicznego Bactrain epoki brązu . Madison w stanie Wisconsin. P. 13. (artykuł przeczytany na konferencji w Azji Południowej)
- Magister Jobling, Tyler-Smith C (sierpień 2003). „Ludzki chromosom Y: ewolucyjny marker dojrzewania”. Recenzje przyrody. Genetyka . 4 (8): 598–612. doi : 10.1038/nrg1124 . PMID 12897772 . S2CID 13508130 .
- Kivisild T , Rootsi S, Metspalu M, Mastana S, Kaldma K, Parik J, et al. (luty 2003). „Dziedzictwo genetyczne pierwszych osadników przetrwało zarówno w populacjach indiańskich, jak i kastowych” . American Journal of Human Genetics . 72 (2): 313–32. doi : 10.1086/346068 . PMC 379225 . PMID 12536373 .
- Metspalu M, Kivisild T, Metspalu E, Parik J, Hudjashov G, Kaldma K, et al. (sierpień 2004). „Większość istniejących granic mtDNA w południowej i południowo-zachodniej Azji została prawdopodobnie ukształtowana podczas początkowego zasiedlania Eurazji przez anatomicznie współczesnego człowieka” . Genetyka BMC . 5 : 26. doi : 10.1186/1471-2156-5-26 . PMC 516768 . PMID 15339343 .
- Patowary A, Purkanti R, Singh M, Chauhan RK, Bhartiya D, Dwivedi OP i in. (lipiec 2012). „Systematyczna analiza i funkcjonalna adnotacja zmian w genomie indyjskiego osobnika”. Mutacja ludzka . 33 (7): 1133–40. doi : 10.1002/humu.22091 . PMID 22461382 . S2CID 11466942 .
- Rootsi S, Magri C, Kivisild T, Benuzzi G, Help H, Bermisheva M, et al. (lipiec 2004). "Filogeografia haplogrupy I chromosomu Y ujawnia odrębne domeny przepływu genów prehistorycznych w Europie" . American Journal of Human Genetics . 75 (1): 128–37. doi : 10.1086/422196 . PMC 1181996 . PMID 15162323 .
- Qamar R, Ayub Q, Mohyuddin A, Helgason A, Mazhar K, Mansoor A, et al. (maj 2002). „Odmiana chromosomu Y DNA w Pakistanie” . American Journal of Human Genetics . 70 (5): 1107-24. doi : 10.1086/339929 . PMC 447589 . PMID 11898125 .
- Semino O, Magri C, Benuzzi G, Lin AA, Al-Zahery N, Battaglia V, et al. (maj 2004). „Pochodzenie, dyfuzja i różnicowanie haplogrup E i J chromosomu Y: wnioski dotyczące neolityzacji Europy i późniejszych wydarzeń migracyjnych w rejonie Morza Śródziemnego” . American Journal of Human Genetics . 74 (5): 1023–34. doi : 10.1086/386295 . PMC 1181965 . PMID 15069642 .
Źródła
- Indyjskie Konsorcjum Odmian Genomu (kwiecień 2008). „Genetyczny krajobraz mieszkańców Indii: płótno do eksploracji genów choroby”. Dziennik Genetyki . 87 (1): 3–20. doi : 10.1007/s12041-008-0002-x . PMID 18560169 . S2CID 21473349 .
- Endicott P, Metspalu M, Kivisild T (2007). „Genetyczne dowody na nowoczesne rozprzestrzenianie się człowieka w Azji Południowej: chromosomy Y i perspektywy mitochondrialnego DNA” . W Michael D. Petraglia, Bridget Allchin (red.). Ewolucja i historia populacji ludzkich w Azji Południowej . Skoczek. s. 201–228. Numer ISBN 978-1-4020-5561-4.
- Hemphill B, Lukacs J, Kennedy K (1991). „Adaptacje biologiczne i powinowactwa Harappanów z epoki brązu”. Na łące RH (red.). Wykopaliska Harappa 1986-1990: multidyscyplinarne podejście do urbanistyki trzeciego tysiąclecia . s. 137-82. Numer ISBN 978-0-9629110-1-9.
- Kennedy'ego KA (1984). „Ponowna ocena teorii pochodzenia rasowego ludzi cywilizacji doliny Indusu z najnowszych danych antropologicznych”. W Kennedy KA, Possehl GL (red.). Studia z archeologii i paleoantropologii Azji Południowej . Atlantic Highlands, NJ : Nauki humanistyczne Press. s. 99–107.
- Kennedy'ego KA (1995). „Czy w prehistorycznych zapisach szkieletowych z Azji Południowej zidentyfikowano Aryjczyków?” . W George Erdosy (red.). Indo-Aryjczycy starożytnej Azji Południowej . Waltera de Gruytera. s. 49–54. Numer ISBN 978-3-11-014447-5.
- Kivisild T (2000b). Początki populacji południowej i zachodniej Eurazji: badanie mtDNA (PDF) . Uniwersytet w Tartu, Estonia. (doktorat)
- Maskarenhas DD, Raina A, Aston CE, Sanghera DK (2015). „Rekonstrukcja genetyczna i kulturowa migracji starożytnego rodowodu” . BioMed Badania Międzynarodowe . 2015 : 651415. doi : 10.1155/2015/651415 . PMC 4605215 . PMID 26491681 .
- Narasimhan VM, Anthony D, Mallory J, Reich D (2018). Formacja genomowa Azji Południowej i Środkowej . bioRxiv 10.1101 /292581 . doi : 10.1101/292581 . hdl : 21.11116/0000-0001-E7B3-0 . S2CID 89658279 .
- Oppenheimera S (2003). The Real Eve: Modern Man's Journey z Afryki . Nowy Jork: Wydawnictwo Carroll i Graf. Numer ISBN 978-0-7867-1192-5.
- Pamjav (grudzień 2012). „Krótka komunikacja: nowe markery binarne chromosomu Y poprawiają rozdzielczość filogenetyczną w obrębie haplogrupy R1a1”. American Journal of Physical Anthropology . 149 (4): 611–615. doi : 10.1002/ajpa.22167 . PMID 23115110 . S2CID 4820868 .
- Renfrew C, Boyle K, wyd. (2000a). Indyjskie pochodzenie: klucz do zrozumienia ludzkiej różnorodności w Europie i poza nią (PDF) . Numer ISBN 978-1-902937-08-3.
- Silva M, Oliveira M, Vieira D, Brandão A, Rito T, Pereira JB, et al. (marzec 2017). „Chronologia genetyczna na subkontynencie indyjskim wskazuje na rozproszenie silnie zorientowane na płeć” . Biologia ewolucyjna BMC . 17 (1): 88. doi : 10.1186/s12862-017-0936-9 . PMC 5364613 . PMID 28335724 .
- Underhill PA (2003). „Wnioskowanie historii ludzkości: wskazówki z haplotypów chromosomu Y”. Sympozja Cold Spring Harbor na temat biologii ilościowej . 68 : 487–93. doi : 10.1101/sqb.2003.68.487 . PMID 15338652 . S2CID 15527457 .
- Underhill PA, Poznik GD, Rootsi S, Järve M, Lin AA, Wang J, et al. (styczeń 2015). „Struktura filogenetyczna i geograficzna haplogrupy R1a chromosomu Y” . European Journal of Human Genetics . 23 (1): 124–31. doi : 10.1038/ejhg.2014.50 . PMC 4266736 . PMID 24667786 .
- Wells S (2003). Podróż człowieka: genetyczna odyseja . Wydawnictwo Uniwersytetu Princeton. Numer ISBN 9780691115320.
Zewnętrzne linki
- Wprowadzenie do haplogrup i haplotypów , Mark A. Jobling, University of Leicester. [1]
- Journey of Man: People of the World , Bradshaw Foundation, we współpracy ze Stephenem Oppenheimerem .
- Indian Genome Variation Database Institute of Genomics and Integrative Biology
- Lista częstotliwości R2
- Malediwy




