Histon - Histone
W biologii , histony są silnie zasadowe białka bogate w lizyny i argininy reszt, które znajdują się w eukariotycznych jąder komórkowych . Działają jak szpule, wokół których nawija się DNA, tworząc jednostki strukturalne zwane nukleosomami . Nukleosomy z kolei są owijane w 30- nanometrowe włókna, które tworzą ciasno upakowaną chromatynę . Histony zapobiegają splątaniu DNA i chronią je przed uszkodzeniem DNA . Ponadto histony odgrywają ważną rolę w regulacji genów i replikacji DNA . Bez histonów rozwinięty DNA w chromosomach byłby bardzo długi. Na przykład, każda ludzka komórka ma około 1,8 metra DNA po całkowitym rozciągnięciu, jednak po nawinięciu na histony długość ta zmniejsza się do około 90 mikrometrów (0,09 mm) włókien chromatyny o średnicy 30 nm.
Istnieje pięć rodzin histonów, które są oznaczone jako H1/H5 (histony łącznika), H2, H3 i H4 (histony rdzeniowe). Rdzeń nukleosomu składa się z dwóch dimerów H2A-H2B i tetrameru H3-H4 . Ścisłe owinięcie DNA wokół histonów jest w dużym stopniu wynikiem przyciągania elektrostatycznego między dodatnio naładowanymi histonami a ujemnie naładowanym fosforanowym szkieletem DNA.
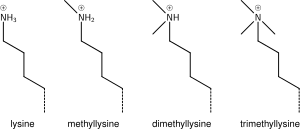
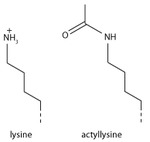
Histony mogą być modyfikowane chemicznie poprzez działanie enzymów regulujących transkrypcję genów. Najczęstszą modyfikacją jest metylacja reszt argininy lub lizyny lub acetylacja lizyny. Metylacja może wpływać na to, jak inne białka, takie jak czynniki transkrypcyjne, oddziałują z nukleosomami. Acetylacja lizyny eliminuje ładunek dodatni lizyny, osłabiając w ten sposób przyciąganie elektrostatyczne między histonem a DNA, co skutkuje częściowym rozwinięciem DNA, czyniąc go bardziej dostępnym dla ekspresji genów.
Klasy i warianty
Istnieje pięć głównych rodzin histonów: H1/H5 , H2A , H2B , H3 i H4 . Histony H2A, H2B, H3 i H4 są znane jako histony rdzeniowe, podczas gdy histony H1/H5 są znane jako histony łącznikowe.
Wszystkie rdzenne histony istnieją jako dimery , które są podobne pod tym względem, że wszystkie posiadają domenę fałdowania histonów: trzy alfa helisy połączone dwiema pętlami. To właśnie ta spiralna struktura pozwala na interakcję między różnymi dimerami, szczególnie w stylu głowa-ogon (zwany również motywem uścisku dłoni). Powstałe cztery różne dimery łączą się, tworząc jeden oktameryczny rdzeń nukleosomowy o średnicy około 63 Angstremów ( cząstka podobna do solenoidu (DNA) ). Około 146 par zasad (pz) DNA owija się wokół tej cząstki rdzenia 1,65 razy w lewoskrętnym superhelikalnym skręcie, dając cząstkę o średnicy około 100 angstremów. Histon łącznikowy H1 wiąże nukleosom w miejscach wejścia i wyjścia DNA, blokując w ten sposób DNA na miejscu i umożliwiając tworzenie struktury wyższego rzędu. Najbardziej podstawową taką formacją jest włókno lub kulki 10 nm na konformacji struny. Obejmuje to owijanie DNA wokół nukleosomów z około 50 parami zasad DNA oddzielającymi każdą parę nukleosomów (określanych również jako DNA łącznika ). Struktury wyższego rzędu obejmują włókno 30 nm (tworzące nieregularny zygzak) i włókno 100 nm, które są strukturami występującymi w normalnych komórkach. Podczas mitozy i mejozy skondensowane chromosomy są składane w wyniku interakcji między nukleosomami a innymi białkami regulatorowymi.
Histony są podzielone na kanonicznych replikacji zależy od histonów, które są wyrażone w fazę S w cyklu komórkowym i niezależna od replikacji wariantów histonów , wyrażane w czasie całego cyklu komórkowego. U zwierząt geny kodujące histony kanoniczne są zazwyczaj zgrupowane wzdłuż chromosomu, nie mają intronów i wykorzystują strukturę pętli pnia na końcu 3' zamiast ogona poliA . Geny kodujące warianty histonów zwykle nie są zgrupowane, mają introny, a ich mRNA są regulowane ogonami poliA. Złożone organizmy wielokomórkowe zazwyczaj mają większą liczbę wariantów histonów zapewniających różnorodne funkcje. Ostatnie dane gromadzą się na temat roli różnych wariantów histonów, podkreślając funkcjonalne powiązania między wariantami i delikatną regulację rozwoju organizmu. Warianty histonów z różnych organizmów, ich klasyfikację i cechy charakterystyczne dla wariantów można znaleźć w bazie danych "HistoneDB 2.0 - Variants" .
Poniżej znajduje się lista ludzkich białek histonowych:
| Super rodzina | Rodzina | Podrodzina | Członkowie |
|---|---|---|---|
| Linker | H1 | H1F | H1F0 , H1FNT , H1FOO , H1FX |
| H1H1 | HIST1H1A , HIST1H1B , HIST1H1C , HIST1H1D , HIST1H1E , HIST1H1T | ||
| Rdzeń | H2A | H2AF | H2AFB1 , H2AFB2 , H2AFB3 , H2AFJ , H2AFV , H2AFX , H2AFY , H2AFY2 , H2AFZ |
| H2A1 | HIST1H2AA , HIST1H2AB , HIST1H2AC , HIST1H2AD , HIST1H2AE , HIST1H2AG , HIST1H2AI , HIST1H2AJ , HIST1H2AK , HIST1H2AL , HIST1H2AM | ||
| H2A2 | HIST2H2AA3 , HIST2H2AC | ||
| H2B | H2BF | H2BFM , H2BFS , H2BFWT | |
| H2B1 | HIST1H2BA , HIST1H2BB , HIST1H2BC , HIST1H2BD , HIST1H2BE , HIST1H2BF , HIST1H2BG , HIST1H2BH , HIST1H2BI , HIST1H2BJ , HIST1H2BK , HIST1H2BL , HIST1H2BM , HIST1H2BN , HIST1H2BO | ||
| H2B2 | HIST2H2BE | ||
| H3 | H3A1 | HIST1H3A , HIST1H3B , HIST1H3C , HIST1H3D , HIST1H3E , HIST1H3F , HIST1H3G , HIST1H3H , HIST1H3I , HIST1H3J | |
| H3A2 | HIST2H3C | ||
| H3A3 | HIST3H3 | ||
| H4 | H41 | HIST1H4A , HIST1H4B , HIST1H4C , HIST1H4D , HIST1H4E , HIST1H4F , HIST1H4G , HIST1H4H , HIST1H4I , HIST1H4J , HIST1H4K , HIST1H4L | |
| H44 | HIST4H4 |
Struktura
Nukleosom rdzeń jest utworzony z dwóch H2A, H2B dimerów i tetramerów H3 H4 tworzących dwie niemal symetryczne połówki za strukturze trzeciorzędowej ( C2 symetrii jednej makromolekuła jest lustrzanym odbiciem drugiej). Dimery H2A-H2B i tetramer H3-H4 również wykazują symetrię pseudodiady. 4 „rdzeniowe” histony (H2A, H2B, H3 i H4) mają stosunkowo podobną strukturę i są wysoce konserwatywne w procesie ewolucji , wszystkie z motywem „ helix turn helix turn helix” (motyw białka wiążącego DNA, który rozpoznaje określoną sekwencję DNA) . Dzielą one również cechę długich "ogonów" na jednym końcu struktury aminokwasowej - jest to lokalizacja modyfikacji potranslacyjnej (patrz poniżej).
Histon Archaeal zawiera jedynie strukturę dimeryczną podobną do H3-H4 wykonaną z tego samego białka. Takie struktury dimeryczne mogą układać się w wysoką superhelisę („hipernukleosom”), na której zwija się DNA w sposób podobny do szpul nukleosomów. Tylko niektóre histony archeonów mają ogony.
Określono, że odległość między szpulami, wokół których komórki eukariotyczne nawijają swoje DNA, wynosi od 59 do 70 Å.
W sumie histony wchodzą w pięć rodzajów interakcji z DNA:
- Mostki solne i wiązania wodorowe między łańcuchami bocznymi aminokwasów zasadowych (zwłaszcza lizyny i argininy ) a tlenami fosforanowymi na DNA
- Dipole helisy tworzą alfa-helisy w H2B, H3 i H4 powodują akumulację dodatniego ładunku netto w punkcie interakcji z ujemnie naładowanymi grupami fosforanowymi na DNA
- Wiązania wodorowe między szkieletem DNA a grupą amidową w głównym łańcuchu białek histonowych
- Niepolarne oddziaływania między cukrami histonowymi i dezoksyrybozowymi na DNA
- Niespecyficzne insercje mniejszych rowków N-końcowych ogonów H3 i H2B w dwa mniejsze rowki w cząsteczce DNA
Wysoce zasadowa natura histonów, poza ułatwianiem interakcji DNA-histon, przyczynia się do ich rozpuszczalności w wodzie.
Histony podlegają potranslacyjnej modyfikacji przez enzymy głównie na ich ogonach N-końcowych, ale także w ich domenach globularnych. Takie modyfikacje obejmują metylację , cytrulinację , acetylację , fosforylację , SUMOilację , ubikwitynację i rybozylację ADP . Wpływa to na ich funkcję regulacji genów.
Ogólnie rzecz biorąc, geny, które są aktywne, mają mniej związanego histonu, podczas gdy nieaktywne geny są silnie związane z histonami podczas interfazy . Wydaje się również, że struktura histonów została zachowana ewolucyjnie , ponieważ wszelkie szkodliwe mutacje byłyby poważnie nieprzystosowane. Wszystkie histony mają wysoce dodatnio naładowany N-koniec z wieloma resztami lizyny i argininy .
Ewolucja i rozmieszczenie gatunków
Histony rdzeniowe znajdują się w jądrach z eukariotycznych komórek iw większości archeowców gromad, ale nie w bakteriach . Jednak histony łącznikowe mają homologi w bakteriach. Wcześniej uważano, że jednokomórkowe glony znane jako bruzdnice są jedynymi eukariontami całkowicie pozbawionymi histonów, jednak późniejsze badania wykazały, że ich DNA nadal koduje geny histonów. W przeciwieństwie do histonów rdzeniowych, bogate w lizynę łącznikowe białka histonowe (H1) znajdują się w bakteriach, inaczej znane jako nukleoproteina HC1/HC2.
Zaproponowano, że białka histonowe są ewolucyjnie spokrewnione z helikalną częścią rozszerzonej domeny AAA+ ATPazy, domeny C i N-końcowej domeny rozpoznającej substrat białek Clp/Hsp100. Pomimo różnic w ich topologii, te trzy fałdy mają homologiczny motyw helisa-nić-helisa (HSH).
Histony archeonów mogą przypominać ewolucyjne prekursory histonów eukariotycznych. Ponadto histony nukleosomu (rdzenia) mogły wyewoluować z białek rybosomalnych ( RPS6 / RPS15 ), z którymi mają wiele wspólnego, będąc zarówno białkami krótkimi, jak i podstawowymi. Białka histonowe należą do najbardziej konserwatywnych białek u eukariontów, co podkreśla ich ważną rolę w biologii jądra. Natomiast dojrzałe plemniki w dużej mierze wykorzystują protaminy do pakowania swojego genomowego DNA, najprawdopodobniej dlatego, że pozwala im to osiągnąć jeszcze wyższy współczynnik pakowania.
Istnieje kilka wariantów formy w niektórych z głównych klas. Mają one homologię sekwencji aminokwasów i podobieństwo strukturalne rdzenia do określonej klasy głównych histonów, ale mają również swoją własną cechę, która różni się od głównych histonów. Te mniejsze histony zwykle pełnią określone funkcje metabolizmu chromatyny. Na przykład, CENPA podobny do histonu H3 jest związany tylko z regionem centromeru chromosomu. Wariant H2A.Z histonu H2A jest powiązany z promotorami genów aktywnie transkrybowanych, a także uczestniczy w zapobieganiu rozprzestrzenianiu się cichej heterochromatyny . Ponadto H2A.Z odgrywa rolę w chromatynie dla stabilności genomu. Inny wariant H2A, H2A.X, jest fosforylowany na S139 w regionach wokół pęknięć dwuniciowych i oznacza region podlegający naprawie DNA . Histon H3.3 jest związany z ciałem genów aktywnie transkrybowanych.
Funkcjonować
Kompaktowanie nici DNA
Histony działają jak szpule, wokół których nawija się DNA. Umożliwia to zagęszczenie niezbędne do dopasowania dużych genomów eukariontów do jąder komórkowych: skompaktowana cząsteczka jest 40 000 razy krótsza niż cząsteczka nieupakowana.
Regulacja chromatyny
Histony przechodzą modyfikacje potranslacyjne, które zmieniają ich interakcję z DNA i białkami jądrowymi. Histony H3 i H4 mają długie ogony wystające z nukleosomu , które w kilku miejscach mogą być modyfikowane kowalencyjnie . Modyfikacje ogona obejmują metylację , acetylację , fosforylację , ubikwitynację , SUMOilację , cytrulinację i rybozylację ADP. Rdzeń histonów H2A i H2B również może być modyfikowany. Uważa się, że kombinacje modyfikacji tworzą kod, tzw. „ kod histonowy ”. Modyfikacje histonów działają w różnych procesach biologicznych, takich jak regulacja genów , naprawa DNA , kondensacja chromosomów ( mitoza ) i spermatogeneza ( mejoza ).
Wspólna nomenklatura modyfikacji histonów to:
- Nazwa histonu (np. H3)
- Jednoliterowy skrót aminokwasu (np. K jak Lysine ) i pozycja aminokwasu w białku
- Rodzaj modyfikacji (Me: metyl , P: fosforan , Ac: acetyl , Ub: ubikwityna )
- Liczba modyfikacji (wiadomo, że tylko Me występuje w więcej niż jednej kopii na resztę. 1, 2 lub 3 to mono-, di- lub tri-metylacja)
Zatem H3K4me1 oznacza monometylację czwartej reszty (lizyny) od początku (tj. N-końca ) białka H3.
| Rodzaj modyfikacji |
Histon | ||||||||
|---|---|---|---|---|---|---|---|---|---|
| H3K4 | H3K9 | H3K14 | H3K27 | H3K79 | H3K36 | H4K20 | H2BK5 | H2BK20 | |
| mono- metylacja | aktywacja | aktywacja | aktywacja | aktywacja | aktywacja | aktywacja | |||
| dimetylacja | represja | represja | aktywacja | ||||||
| tri-metylacja | aktywacja | represja | represja | aktywizacja, represje |
aktywacja | represja | |||
| acetylacja | aktywacja | aktywacja | aktywacja | aktywacja | aktywacja | ||||
Modyfikacja
Opisano ogromny katalog modyfikacji histonów, ale nadal brakuje funkcjonalnego zrozumienia większości. Łącznie uważa się, że modyfikacje histonów mogą leżeć u podstaw kodu histonów , przy czym kombinacje modyfikacji histonów mają określone znaczenie. Jednak większość danych funkcjonalnych dotyczy pojedynczych znaczących modyfikacji histonów, które są biochemicznie podatne na szczegółowe badania.
Chemia
Metylacja lizyny
Dodanie jednej, dwóch lub wielu grup metylowych do lizyny ma niewielki wpływ na chemię histonu; Metylacja pozostawia nienaruszony ładunek lizyny i dodaje minimalną liczbę atomów, więc oddziaływania steryczne są w większości nienaruszone. Jednak białka zawierające m.in. domeny Tudor, chromo lub PHD potrafią rozpoznawać metylację lizyny z wyjątkową czułością i różnicować mono, di i tri-metylolizynę do tego stopnia, że dla niektórych lizyn (np. H4K20) mono, di i tri -metylacja wydaje się mieć różne znaczenia. Z tego powodu metylacja lizyny wydaje się być bardzo pouczającym wskaźnikiem i dominuje w znanych funkcjach modyfikacji histonów.
Serotonylacja glutaminy
Ostatnio wykazano, że dodanie grupy serotoninowej do pozycji 5 glutaminy H3 zachodzi w komórkach serotoninergicznych, takich jak neurony. Jest to część różnicowania komórek serotoninergicznych. Ta modyfikacja potranslacyjna zachodzi w połączeniu z modyfikacją H3K4me3. Serotonylacja wzmaga wiązanie ogólnego czynnika transkrypcyjnego TFIID do kasety TATA .
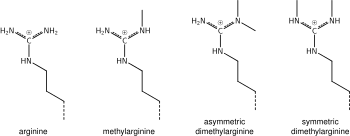
Metylacja argininy
To, co zostało powiedziane powyżej o chemii metylacji lizyny, dotyczy również metylacji argininy, a niektóre domeny białkowe - np. domeny Tudora - mogą być specyficzne dla metyloargininy zamiast metylolizyny. Wiadomo, że arginina jest mono- lub dimetylowana, a metylacja może być symetryczna lub asymetryczna, potencjalnie o różnych znaczeniach.
Cytrulinacja argininy
Enzymy zwane deiminazami peptydyloargininowymi (PAD) hydrolizują grupę iminową argininy i przyłączają grupę ketonową, dzięki czemu na reszcie aminokwasowej jest jeden ładunek dodatni mniej. Proces ten był zaangażowany w aktywację ekspresji genów poprzez osłabienie wiązania zmodyfikowanych histonów z DNA, a tym samym zwiększenie dostępności chromatyny. PAD mogą również wywoływać odwrotny efekt, usuwając lub hamując monometylację reszt argininy na histonach, a tym samym antagonizując pozytywny wpływ metylacji argininy na aktywność transkrypcyjną.
acetylacja lizyny
Dodanie grupy acetylowej ma duży wpływ chemiczny na lizynę, ponieważ neutralizuje ładunek dodatni. Zmniejsza to przyciąganie elektrostatyczne między histonem a ujemnie naładowanym szkieletem DNA, rozluźniając strukturę chromatyny; wysoce acetylowane histony tworzą bardziej dostępną chromatynę i mają tendencję do wiązania się z aktywną transkrypcją. Acetylacja lizyny wydaje się mieć mniej precyzyjne znaczenie niż metylacja, ponieważ acetylotransferazy histonowe mają tendencję do działania na więcej niż jedną lizynę; przypuszczalnie odzwierciedla to potrzebę zmiany wielu lizyn, aby mieć znaczący wpływ na strukturę chromatyny. Modyfikacja obejmuje H3K27ac .
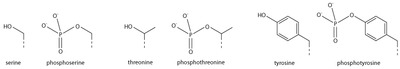
Fosforylacja seryny/treoniny/tyrozyny
Dodanie ujemnie naładowanej grupy fosforanowej może prowadzić do poważnych zmian w strukturze białka, prowadząc do dobrze scharakteryzowanej roli fosforylacji w kontrolowaniu funkcji białka. Nie jest jasne, jakie strukturalne implikacje ma fosforylacja histonów, ale fosforylacja histonów pełni wyraźne funkcje jako modyfikacja potranslacyjna i scharakteryzowano domeny wiążące, takie jak BRCT.
Wpływ na transkrypcję
Większość dobrze zbadanych modyfikacji histonów jest zaangażowana w kontrolę transkrypcji.
Aktywnie transkrybowane geny
Z aktywną transkrypcją szczególnie związane są dwie modyfikacje histonów:
- Trimetylacja H3 lizyny 4 (H3K4me3)
- Ta trimetylacja zachodzi na promotorze aktywnych genów i jest realizowana przez kompleks COMPASS . Pomimo zachowania tego kompleksu i modyfikacji histonów z drożdży na ssaki, nie jest do końca jasne, jaką rolę odgrywa ta modyfikacja. Jest jednak doskonałym wyznacznikiem aktywnych promotorów, a poziom tej modyfikacji histonów w promotorze genu jest szeroko skorelowany z aktywnością transkrypcyjną genu. Powstanie tego znaku jest powiązane z transkrypcją w dość zawiły sposób: we wczesnej fazie transkrypcji genu polimeraza RNA II przechodzi zmianę z inicjowania na „wydłużanie” , zaznaczoną zmianą stanów fosforylacji polimerazy RNA II C domena terminala (CTD) . Ten sam enzym, który fosforyluje CTD, fosforyluje również kompleks Rad6, który z kolei dodaje znak ubikwityny do H2B K123 (K120 u ssaków). H2BK123Ub występuje w transkrybowanych regionach, ale ten znak jest wymagany, aby COMPASS trimetylował H3K4 na promotorach.
- Trimetylacja lizyny H3 36 ( H3K36me3 )
- Ta trimetylacja zachodzi w ciele aktywnych genów i jest odkładana przez zestaw 2 metylotransferazy. Białko to wiąże się z wydłużającą się polimerazą RNA II , a H3K36Me3 wskazuje na aktywne transkrypcje genów. H3K36Me3 jest rozpoznawany przez kompleks deacetylazy histonowej Rpd3, który usuwa modyfikacje acetylowe z otaczających histonów, zwiększając zagęszczenie chromatyny i hamując fałszywą transkrypcję. Zwiększone zagęszczenie chromatyny uniemożliwia czynnikom transkrypcyjnym dostęp do DNA i zmniejsza prawdopodobieństwo zainicjowania nowych zdarzeń transkrypcyjnych w ciele genu. Proces ten pomaga zatem zapewnić, że transkrypcja nie zostanie przerwana.
Wyparte geny
Trzy modyfikacje histonów są szczególnie związane z represjonowanymi genami:
- Trimetylacja lizyny H3 27 (H3K27me3)
- Ta modyfikacja histonów jest osadzana przez Polycomb złożonej PRC2. Jest wyraźnym markerem represji genów i prawdopodobnie jest wiązany przez inne białka, aby wywierać funkcję represyjną. Inny kompleks polycomb , PRC1, może wiązać H3K27me3 i dodaje modyfikację histonową H2AK119Ub, która wspomaga zagęszczanie chromatyny. Na podstawie tych danych wydaje się, że PRC1 jest rekrutowana przez działanie PRC2, jednak ostatnie badania pokazują, że PRC1 jest rekrutowana do tych samych miejsc przy braku PRC2.
- Di i tri-metylacja H3 lizyny 9 (H3K9me2/3)
- H3K9me2/3 jest dobrze scharakteryzowanym markerem heterochromatyny i dlatego jest silnie związany z represją genów. Tworzenie heterochromatyny najlepiej zbadano u drożdży Schizosaccharomyces pombe , gdzie jest inicjowane przez rekrutację kompleksu wyciszania transkrypcji indukowanego przez RNA (RITS) do dwuniciowych RNA wytworzonych z powtórzeń centromerowych . RITS rekrutuje metylotransferazę histonową Clr4, która odkłada H3K9me2/3. Proces ten nazywa się metylacją histonów . H3K9Me2/3 służy jako miejsce wiązania dla rekrutacji Swi6 ( białko heterochromatyny 1 lub HP1, inny klasyczny marker heterochromatyny), który z kolei rekrutuje dalsze aktywności represyjne, w tym modyfikatory histonów, takie jak deacetylazy histonów i metylotransferazy histonów .
- Trimetylacja lizyny H4 20 ( H4K20me 3)
- Ta modyfikacja jest ściśle związana z heterochromatyną, chociaż jej znaczenie funkcjonalne pozostaje niejasne. Znak ten jest umieszczany przez metylotransferazę Suv4-20h, która jest przynajmniej częściowo rekrutowana przez białko heterochromatyny 1 .
Promotory biwalentne
Analiza modyfikacji histonów w embrionalnych komórkach macierzystych (i innych komórkach macierzystych) ujawniła wiele promotorów genów niosących zarówno H3K4Me3, jak i H3K27Me3 , innymi słowy, promotory te wykazują jednocześnie zarówno znaki aktywujące, jak i represjonujące. Ta osobliwa kombinacja modyfikacji oznacza geny, które są gotowe do transkrypcji; nie są wymagane w komórkach macierzystych, ale są szybko potrzebne po zróżnicowaniu w niektóre linie. Gdy komórka zaczyna się różnicować, te dwuwartościowe promotory są rozdzielane do stanów aktywnych lub represyjnych, w zależności od wybranej linii.
Inne funkcje
Uszkodzenie DNA
Oznaczanie miejsc uszkodzeń DNA jest ważną funkcją modyfikacji histonów. Chroni również DNA przed zniszczeniem przez ultrafioletowe promieniowanie słoneczne.
- Fosforylacja H2AX w serynie 139 (γH2AX)
- Fosforylowany H2AX (znany również jako gamma H2AX) jest markerem pęknięć podwójnej nici DNA i stanowi część odpowiedzi na uszkodzenie DNA . H2AX ulega fosforylacji wcześnie po wykryciu pęknięcia podwójnej nici DNA i tworzy domenę rozciągającą się na wiele kilozasad po obu stronach uszkodzenia. Gamma H2AX działa jako miejsce wiązania białka MDC1, które z kolei rekrutuje kluczowe białka naprawcze DNA (ten złożony temat jest dobrze omówiony) i jako taki, gamma H2AX stanowi istotną część mechanizmu zapewniającego stabilność genomu.
- Acetylowanie lizyny H3 56 (H3K56Ac)
- H3K56Acx jest wymagany do stabilności genomu. H3K56 jest acetylowany przez kompleks p300/Rtt109, ale jest szybko deacetylowany wokół miejsc uszkodzenia DNA. Acetylacja H3K56 jest również wymagana do ustabilizowania zablokowanych widełek replikacyjnych, zapobiegając niebezpiecznemu zawaleniu się widełek replikacyjnych. Chociaż ogólnie ssaki znacznie częściej wykorzystują modyfikacje histonów niż mikroorganizmy, główna rola H3K56Ac w replikacji DNA występuje tylko u grzybów, co stało się celem rozwoju antybiotyków.
Naprawa DNA
- Trimetylacja lizyny H3 36 (H3K36me3)
H3K36me3 ma zdolność rekrutacji kompleksu MSH2-MSH6 (hMutSα) szlaku naprawy niedopasowania DNA . Konsekwentnie, regiony ludzkiego genomu o wysokim poziomie H3K36me3 gromadzą mniej mutacji somatycznych z powodu aktywności naprawy niedopasowania .
Kondensacja chromosomów
- Fosforylacja H3 w serynie 10 (fosfo-H3S10)
- Mitotyczna kinaza aurora B fosforyluje histon H3 w serynie 10, wywołując kaskadę zmian, które pośredniczą w kondensacji chromosomów mitotycznych. Chromosomy skondensowane wybarwiają się zatem bardzo silnie pod kątem tego znaku, ale fosforylacja H3S10 jest również obecna w niektórych miejscach chromosomu poza mitozą, na przykład w obwodowej heterochromatynie komórek podczas G2. Fosforylacja H3S10 została również powiązana z uszkodzeniem DNA powodowanym przez tworzenie pętli R w miejscach o wysokim stopniu transkrypcji.
- Fosforylacja H2B w serynie 10/14 (fosfo-H2BS10/14)
- Fosforylacja H2B w serynie 10 (drożdże) lub serynie 14 (ssaki) jest również powiązana z kondensacją chromatyny, ale w zupełnie innym celu pośredniczenia w kondensacji chromosomów podczas apoptozy. Ten znak nie jest po prostu późnym działaniem przypadkowym w apoptozie, ponieważ drożdże niosące mutacje tej reszty są odporne na apoptotyczną śmierć komórek wywołaną nadtlenkiem wodoru.
Nałóg
Modyfikacje epigenetyczne ogonów histonów w określonych obszarach mózgu mają kluczowe znaczenie w uzależnieniach. Gdy wystąpią określone zmiany epigenetyczne, wydają się one być długotrwałymi „bliznami molekularnymi”, które mogą odpowiadać za utrzymywanie się uzależnień.
Palacze papierosów (około 15% populacji USA) są zwykle uzależnieni od nikotyny . Po 7 dniach leczenia myszy nikotyną, acetylacja histonu H3 i histonu H4 wzrosła na promotorze FosB w jądrze półleżącym mózgu, powodując 61% wzrost ekspresji FosB. Zwiększyłoby to również ekspresję wariantu splicingowego Delta FosB . W jądrze półleżącym mózgu Delta FosB działa jako „podtrzymywany przełącznik molekularny” i „główne białko kontrolne” w rozwoju uzależnienia .
Około 7% populacji USA jest uzależnionych od alkoholu . U szczurów narażonych na działanie alkoholu przez okres do 5 dni obserwowano wzrost acetylacji lizyny 3 histonu 9 w pronocyceptynie w kompleksie ciała migdałowatego mózgu . Ta acetylacja jest aktywującym znacznikiem dla pronocyceptyny. Układ receptorów opioidowych nocyceptyny/nocyceptyny bierze udział we wzmacnianiu lub kondycjonowaniu działania alkoholu.
Uzależnienie od metamfetaminy występuje u około 0,2% populacji USA. Przewlekłe metamfetaminy powoduje metylację lizyny w pozycji 4 histonu 3, umieszczonego na promotorów tych c-fos i receptorów CC chemokiny 2 (CCR2) genów aktywacji tych genów w jądrze półleżącym (NAC). Powszechnie wiadomo, że c-fos odgrywa ważną rolę w uzależnieniu . CCR2 gen jest również ważne w uzależnienia od mutacji inaktywacji tego nałogu upośledza genowych.
Synteza
Pierwszym etapem powielania struktury chromatyny jest synteza białek histonowych: H1, H2A, H2B, H3, H4. Białka te są syntetyzowane podczas fazy S cyklu komórkowego. Istnieją różne mechanizmy, które przyczyniają się do zwiększenia syntezy histonów.
Drożdże
Drożdże niosą jedną lub dwie kopie każdego genu histonowego, które nie są skupione, ale raczej rozproszone w chromosomach. Transkrypcja genów histonów jest kontrolowana przez wiele białek regulatorowych genów, takich jak czynniki transkrypcyjne, które wiążą się z regionami promotora histonów. W pączkujących drożdżach genem kandydującym do aktywacji ekspresji genu histonów jest SBF. SBF jest czynnikiem transkrypcyjnym, który jest aktywowany w późnej fazie G1, kiedy dysocjuje od swojego represora Whi5 . Dzieje się tak, gdy Whi5 jest fosforylowany przez Cdc8, który jest G1/S Cdk. Tłumienie ekspresji genów histonów poza fazami S zależy od białek Hir, które tworzą nieaktywną strukturę chromatyny w locus genów histonów, powodując blokowanie aktywatorów transkrypcji.
Metazoan
W metazoanach wzrost szybkości syntezy histonów wynika ze zwiększenia przetwarzania pre-mRNA do jego dojrzałej postaci oraz zmniejszenia degradacji mRNA; powoduje to wzrost aktywnego mRNA do translacji białek histonowych. Stwierdzono, że mechanizm aktywacji mRNA polega na usunięciu segmentu końca 3' nici mRNA i jest zależny od połączenia z białkiem wiążącym pień-pętlę ( SLBP ). SLBP stabilizuje również mRNA histonów podczas fazy S poprzez blokowanie degradacji przez 3'hExo-nukleazę. Poziomy SLBP są kontrolowane przez białka cyklu komórkowego, powodując akumulację SLBP, gdy komórki wchodzą w fazę S i degradację, gdy komórki opuszczają fazę S. SLBP są oznaczone do degradacji przez fosforylację dwóch reszt treoniny przez kinazy zależne od cykliny, prawdopodobnie cyklinę A/cdk2, na końcu fazy S. Metazoans mają również wiele kopii genów histonowych skupionych na chromosomach, które są zlokalizowane w strukturach zwanych ciałami Cajala, jak określono za pomocą analizy wychwytywania konformacji chromosomów całego genomu (4C-Seq).
Związek między kontrolą cyklu komórkowego a syntezą
Białko jądrowe ataksja-teleangiektazja (NPAT), znane również jako koaktywator białka jądrowego transkrypcji histonów, jest czynnikiem transkrypcyjnym, który aktywuje transkrypcję genu histonów na chromosomach 1 i 6 ludzkich komórek. NPAT jest również substratem cykliny E-Cdk2, która jest wymagana do przejścia między fazą G1 a fazą S. NPAT aktywuje ekspresję genu histonów dopiero po jego ufosforylowaniu przez cyklinę G1/S-Cdk E-Cdk2 we wczesnej fazie S. Wskazuje to na ważne powiązanie regulacyjne między kontrolą cyklu komórkowego a syntezą histonów.
Historia
Histony zostały odkryte w 1884 roku przez Albrechta Kossla . Słowo „histon” pochodzi z końca XIX wieku i wywodzi się od niemieckiego słowa „Histon” , które samo w sobie ma niepewne pochodzenie, być może od starożytnej greki ἵστημι (hístēmi, „postawić się”) lub ἱστός (histós, „krosno” ).
We wczesnych latach sześćdziesiątych, zanim poznano typy histonów i zanim okazało się, że histony są wysoce konserwatywne w organizmach zróżnicowanych taksonomicznie, James F. Bonner i jego współpracownicy rozpoczęli badania tych białek, o których wiadomo było, że są ściśle związane z DNA w jądro organizmów wyższych. Bonner i jego kolega podoktorancki Ru Chih C. Huang wykazali, że wyizolowana chromatyna nie będzie wspierać transkrypcji RNA w probówce, ale jeśli histony zostaną wyekstrahowane z chromatyny, RNA może być transkrybowane z pozostałego DNA. Ich artykuł stał się klasykiem cytowań. Paul T'so i James Bonner zwołali Światowy Kongres Chemii i Biologii Histonów w 1964 roku, na którym stało się jasne, że nie ma zgody co do liczby rodzajów histonów i nikt nie wie, jak będą się one porównywać w izolacji od różne organizmy. Bonner i jego współpracownicy opracowali następnie metody oddzielania każdego typu histonów, oczyścili poszczególne histony, porównali składy aminokwasowe tego samego histonu z różnych organizmów i porównali sekwencje aminokwasowe tego samego histonu z różnych organizmów we współpracy z Emilem Smithem z UCLA. Na przykład odkryli, że sekwencja histonu IV jest wysoce konserwatywna między groszkiem a grasicą cielęcą. Jednak ich prace nad charakterystyką biochemiczną poszczególnych histonów nie ujawniły, w jaki sposób histony oddziałują ze sobą ani z DNA, z którym były ściśle związane.
Również w latach 60. Vincent Allfrey i Alfred Mirsky sugerowali, na podstawie swoich analiz histonów, że acetylacja i metylacja histonów może zapewnić mechanizm kontroli transkrypcji, ale nie mieli dostępnej szczegółowej analizy, którą późniejsi badacze byli w stanie przeprowadzić. aby pokazać, w jaki sposób taka regulacja może być specyficzna dla genu. Do wczesnych lat 90. histony były przez większość odrzucane jako obojętny materiał pakujący dla eukariotycznego DNA jądrowego, co częściowo opierało się na modelach Marka Ptashne'a i innych, którzy wierzyli, że transkrypcja jest aktywowana przez interakcje białko-DNA i białko-białko w dużej mierze nagich matryc DNA, jak to ma miejsce w przypadku bakterii.
W latach 80. Yahli Lorch i Roger Kornberg wykazali, że nukleosom na promotorze rdzenia zapobiega inicjacji transkrypcji in vitro, a Michael Grunstein wykazał, że histony hamują transkrypcję in vivo, prowadząc do koncepcji nukleosomu jako ogólnego represora genów. Uważa się, że zwolnienie z represji obejmuje zarówno modyfikację histonów, jak i działanie kompleksów remodelujących chromatynę. Vincent Allfrey i Alfred Mirsky wcześniej proponowali rolę modyfikacji histonów w aktywacji transkrypcyjnej, uważanej za molekularną manifestację epigenetyki. Michael Grunstein i David Allis znaleźli poparcie dla tej propozycji w znaczeniu acetylacji histonów dla transkrypcji w drożdżach i aktywności aktywatora transkrypcji Gcn5 jako acetylotransferazy histonowej.
Odkrycie histonu H5 wydaje sięgają 1970 roku i jest obecnie uznawana za izoformy z histonu H1 .