Onkogenomika - Oncogenomics
Onkogenomika to poddziedzina genomiki, która charakteryzuje geny związane z rakiem . Koncentruje się na zmianach genomicznych, epigenomicznych i transkrypcyjnych w raku.
Rak jest chorobą genetyczną spowodowaną nagromadzeniem mutacji DNA i zmian epigenetycznych prowadzących do niepohamowanej proliferacji komórek i powstawania nowotworów . Celem onkogenomiki jest identyfikacja nowych onkogenów lub genów supresorowych nowotworów, które mogą zapewnić nowy wgląd w diagnostykę raka, przewidywanie wyników klinicznych nowotworów i nowych celów terapii przeciwnowotworowych. Sukces ukierunkowanych terapii przeciwnowotworowych, takich jak Gleevec , Herceptin i Avastin, wzbudził nadzieje dla onkogenomiki na wyjaśnienie nowych celów w leczeniu raka.
Poza zrozumieniem podstawowych mechanizmów genetycznych, które inicjują lub napędzają progresję raka, onkogenomika ma na celu spersonalizowane leczenie raka. Rak rozwija się z powodu mutacji DNA i zmian epigenetycznych, które gromadzą się losowo. Identyfikacja i ukierunkowanie mutacji u pojedynczego pacjenta może prowadzić do zwiększenia skuteczności leczenia.
Zakończenie projektu Human Genome Project ułatwiło rozwój onkogenomiki i zwiększyło możliwości badaczy w znajdowaniu onkogenów. W badaniach onkogenomicznych zastosowano technologie sekwencjonowania i globalne techniki profilowania metylacji.
Historia
Era genomiki rozpoczęła się w latach dziewięćdziesiątych, wraz z generowaniem sekwencji DNA wielu organizmów. W XXI wieku zakończenie projektu Human Genome Project umożliwiło badanie genomiki funkcjonalnej i badanie genomów nowotworowych. Rak jest głównym celem.
Era epigenomiki w dużej mierze rozpoczęła się niedawno, około 2000 roku. Jednym z głównych źródeł zmian epigenetycznych jest zmieniona metylacja wysp CpG w regionie promotorowym genów (patrz metylacja DNA w raku ). Szereg niedawno opracowanych metod pozwala ocenić stan metylacji DNA w nowotworach w porównaniu z normalnymi tkankami. Niektóre metody oceniają metylację CpG zlokalizowanych w różnych klasach loci, w tym na wyspach CpG, brzegach i półkach, a także promotorach, ciałach genów i regionach międzygenowych. Rak jest również głównym przedmiotem badań epigenetycznych.
Dostęp do sekwencjonowania całego genomu raka jest ważny dla badań nad rakiem (lub genomem raka), ponieważ:
- Mutacje są bezpośrednią przyczyną raka i określają fenotyp nowotworu .
- Dostęp do próbek tkanek nowotworowych i normalnych od tego samego pacjenta oraz fakt, że większość mutacji nowotworowych reprezentuje zdarzenia somatyczne , pozwalają na identyfikację mutacji specyficznych dla nowotworu.
- Mutacje nowotworowe kumulują się i czasami są związane ze stadium choroby. Rozróżnia się przerzuty i lekooporność.
Dostęp do profilowania metylacji jest ważny dla badań nad rakiem, ponieważ:
- Sterowniki epi, wraz ze sterownikami Mut, mogą działać jako bezpośrednie przyczyny nowotworów
- Epimutacje raka mają charakter kumulacyjny i czasami są związane ze stadium choroby
Sekwencjonowanie całego genomu
Pierwszy genom nowotworowy został zsekwencjonowany w 2008 roku. W tym badaniu zsekwencjonowano typowy genom ostrej białaczki szpikowej (AML) i jego normalny odpowiednik genomowy uzyskany od tego samego pacjenta. Porównanie ujawniło dziesięć zmutowanych genów. Dwie były już uważane za przyczyniające się do progresji nowotworu: wewnętrzną duplikację tandem z FLT3 receptora kinazy tyrozynowej genu, który aktywuje kinazę sygnalizację i wiąże się ze złym rokowaniem, a wstawienie czterech zasad w eksonie 12 NPM1 genu (NPMc). Mutacje te znajdują się w 25-30% guzów AML i uważa się, że przyczyniają się one do progresji choroby, a nie powodują ją bezpośrednio.
Pozostałe 8 to nowe mutacje i wszystkie były zmianami pojedynczej zasady: cztery były w rodzinach silnie związanych z patogenezą raka ( PTPRT , CDH24, PCLKC i SLC15A1 ). Pozostałe cztery nie miały wcześniejszego związku z patogenezą raka. Miały potencjalne funkcje w szlakach metabolicznych, które sugerowały mechanizmy, dzięki którym mogą działać na rzecz promocji raka (KNDC1, GPR124 , EB12, GRINC1B)
Geny te są zaangażowane w szlaki, o których wiadomo, że przyczyniają się do patogenezy raka, ale przed tym badaniem większość z nich nie byłaby kandydatami do ukierunkowanej terapii genowej. Analiza ta potwierdziła podejście sekwencjonowania całego genomu raka w identyfikacji mutacji somatycznych oraz znaczenie równoległego sekwencjonowania genomów normalnych i nowotworowych komórek.
W 2011 roku zsekwencjonowano genom wyjątkowego pacjenta z rakiem pęcherza moczowego, którego guz został wyeliminowany przez lek ewerolimus , ujawniając mutacje w dwóch genach, TSC1 i NF2 . Mutacje rozregulowały mTOR , białko hamowane przez ewerolimus, umożliwiając mu nieograniczoną reprodukcję. W efekcie w 2015 roku w Narodowym Instytucie Raka powstała Inicjatywa Wyjątkowych Respondentów. Inicjatywa umożliwia takim wyjątkowym pacjentom (którzy reagowali pozytywnie przez co najmniej sześć miesięcy na lek przeciwnowotworowy, który zwykle nie działa), sekwencjonowanie ich genomów w celu zidentyfikowania odpowiednich mutacji. Po zidentyfikowaniu, inni pacjenci mogliby zostać przebadani pod kątem tych mutacji, a następnie otrzymać lek. W 2016 r. W tym celu w 2015 r. rozpoczęto ogólnokrajowe badanie leków przeciwnowotworowych, obejmujące nawet dwadzieścia czterysta ośrodków. Pacjentom z odpowiednimi mutacjami dopasowuje się jeden z ponad czterdziestu leków.
W 2014 roku Centrum Onkologii Molekularnej wprowadziło test MSK-IMPACT, narzędzie przesiewowe, które szuka mutacji w 341 genach związanych z rakiem. Do 2015 roku przebadano ponad pięć tysięcy pacjentów. Pacjenci z odpowiednimi mutacjami kwalifikują się do udziału w badaniach klinicznych, które zapewniają terapię celowaną.
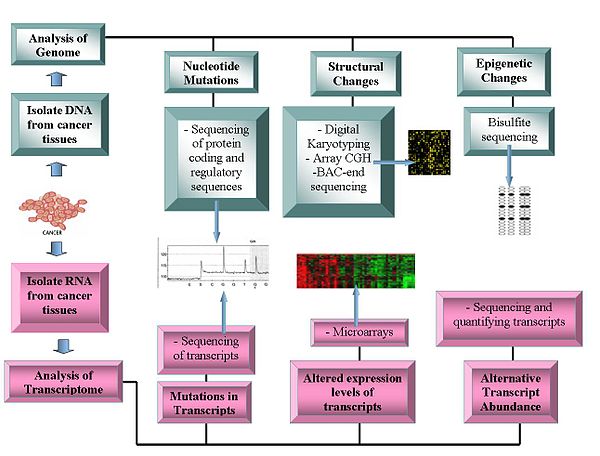
Technologie
Technologie genomiczne obejmują:
Sekwencjonowanie genomu
- Sekwencjonowanie DNA :sekwencery oparte na pirosekwencjonowaniu oferują stosunkowo tanią metodę generowania danych sekwencyjnych.
- Hybrydyzacja porównawcza genomu z macierzą : Ta technika mierzyróżnice w liczbie kopii DNA między genomem normalnym a nowotworowym. Wykorzystuje intensywność fluorescencji z próbek znakowanych fluorescencyjnie, które są hybrydyzowane ze znanymi sondami na mikromacierzy.
- Reprezentacyjna analiza mikromacierzy oligonukleotydów : Wykrywa zmienność liczby kopii przy użyciu amplifikowanych fragmentów genomowych poddanych trawieniu restrykcyjnie, które są hybrydyzowane z ludzkimi oligonukleotydami, osiągając rozdzielczość między 30 a 35 kbit/s.
- Cyfrowe Kariotypowanie : Wykrywa zmienność liczby kopii za pomocą znaczników genomicznych uzyskanych w wyniku trawienia enzymami restrykcyjnymi . Znaczniki te są następnie łączone w znaczniki, łączone, klonowane, sekwencjonowane i mapowane z powrotem do genomu referencyjnego w celu oceny gęstości znaczników.
- Sekwencjonowanie końców sztucznych chromosomów bakteryjnych (BAC) ( profilowanie sekwencji końcowych ) : Identyfikuje miejscaprzerwania chromosomów poprzez generowanie biblioteki BAC z genomu raka i sekwencjonowanie ich końców. Klony BAC, które zawierają aberracje chromosomowe, mają sekwencje końcowe, które nie mapują się do podobnego regionu genomu referencyjnego, identyfikując w ten sposób punkt złamania chromosomu.
Transkryptomy
- Mikromacierze : Oceń obfitość transkrypcji . Przydatne w klasyfikacji, prognozowaniu, podnoszą możliwości różnicowania metod leczenia i wspomagają identyfikację mutacji w regionach kodujących białka. Względna obfitość alternatywnych transkryptów stała się ważną cechą badań nad rakiem. Poszczególne alternatywne formy transkryptu korelują z określonymi typami raka.
- sekwencja RNA
Bioinformatyka i analiza funkcjonalna onkogenów
Technologie bioinformatyczne umożliwiają analizę statystyczną danych genomowych. Funkcjonalne cechy onkogenów nie zostały jeszcze ustalone. Potencjalne funkcje obejmują ich zdolności transformacyjne związane z powstawaniem guza i określone role na każdym etapie rozwoju raka.
Po wykryciu somatycznych mutacji nowotworowych w kohorcie próbek nowotworowych można przeprowadzić bioinformatyczne analizy obliczeniowe w celu zidentyfikowania prawdopodobnych funkcjonalnych i prawdopodobnych mutacji kierujących. Istnieją trzy główne podejścia rutynowo stosowane do tej identyfikacji: mapowanie mutacji, ocena wpływu mutacji funkcji białka lub elementu regulacyjnego i znajdowanie oznak pozytywnej selekcji w kohorcie nowotworów. Podejścia niekoniecznie są sekwencyjne, jednak istnieją ważne relacje pierwszeństwa między elementami z różnych podejść. Na każdym etapie używane są różne narzędzia.
Operomika
Operomika ma na celu zintegrowanie genomiki, transkryptomiki i proteomiki, aby zrozumieć mechanizmy molekularne leżące u podstaw rozwoju raka.
Onkogenomika porównawcza
Onkogenomika porównawcza wykorzystuje porównania międzygatunkowe w celu identyfikacji onkogenów. Badania te obejmują badanie genomów, transkryptomów i proteomów raka w organizmach modelowych, takich jak myszy, identyfikowanie potencjalnych onkogenów i odwoływanie się do próbek raka ludzkiego, aby sprawdzić, czy homologi tych onkogenów są ważne w wywoływaniu nowotworów u ludzi. Zmiany genetyczne w modelach mysich są podobne do tych występujących w ludzkich nowotworach. Modele te są generowane metodami obejmującymi retrowirusową mutagenezę insercyjną lub przeszczep komórek rakowych.
Źródło mutacji sterowników nowotworowych, mutageneza nowotworowa
Mutacje stanowią surowiec do doboru naturalnego w ewolucji i mogą być spowodowane błędami replikacji DNA, działaniem egzogennych mutagenów lub endogennym uszkodzeniem DNA. Maszyneria replikacji i utrzymywania genomu może zostać uszkodzona przez mutacje lub zmieniona przez warunki fizjologiczne i zróżnicowane poziomy ekspresji w raku (patrz odniesienia w ).
Jak wskazano przez Gao i wsp., stabilność i integralność ludzkiego genomu jest utrzymywana przez system odpowiedzi na uszkodzenie DNA (DDR). Nienaprawione uszkodzenie DNA jest główną przyczyną mutacji, które napędzają karcynogenezę. Jeśli naprawa DNA jest niewystarczająca, uszkodzenia DNA mają tendencję do kumulacji. Takie nadmierne uszkodzenie DNA może zwiększyć błędy mutacyjne podczas replikacji DNA z powodu podatnej na błędy syntezy translekcji . Nadmierne uszkodzenie DNA może również zwiększyć zmiany epigenetyczne z powodu błędów podczas naprawy DNA. Takie mutacje i zmiany epigenetyczne mogą powodować raka . Geny DDR są często tłumione w ludzkim raku przez mechanizmy epigenetyczne. Taka represja może obejmować metylację DNA regionów promotora lub represję genów DDR przez mikroRNA. Represja epigenetyczna genów DDR występuje częściej niż mutacja genów w wielu typach raka (patrz Epigenetyka nowotworów ). Tak więc represje epigenetyczne często odgrywają ważniejszą rolę niż mutacja w zmniejszaniu ekspresji genów DDR. Ta zmniejszona ekspresja genów DDR jest prawdopodobnie ważnym motorem kancerogenezy.
Kontekst sekwencji nukleotydowej wpływa na prawdopodobieństwo mutacji, a analiza mutacyjnych (zmiennych) motywów DNA może być niezbędna do zrozumienia mechanizmów mutagenezy w raku. Takie motywy reprezentują odciski palców interakcji między DNA a mutagenami, między DNA a enzymami naprawy/replikacji/modyfikacji. Przykładami motywów są motyw AID WRCY/RGYW (W = A lub T, R = puryna i Y = pirymidyna) z mutacjami C do T/G/A oraz podatne na błędy mutacje DNA przypisywane pol η związane z AID (od A do G/C/G) w motywach WA/TW.
Inny (agnostyczny) sposób analizy obserwowanych widm mutacji i kontekstu sekwencji DNA mutacji w guzach polega na połączeniu wszystkich mutacji różnych typów i kontekstów z próbek raka w dyskretną dystrybucję. Jeśli dostępnych jest wiele próbek raka, ich mutacje zależne od kontekstu mogą być reprezentowane w postaci nieujemnej macierzy. Matryca ta może być dalej rozłożona na składniki (sygnatury mutacji), które idealnie powinny opisywać poszczególne czynniki mutagenne. Do rozwiązania tego problemu dekompozycji zaproponowano kilka metod obliczeniowych. Pierwsza implementacja metody Non-negative Matrix Factorization (NMF) jest dostępna w Sanger Institute Mutational Signature Framework w postaci pakietu MATLAB. Z drugiej strony, jeśli mutacje z pojedynczej próbki guza są dostępne tylko, pakiet DeconstructSigs R i serwer MutaGene mogą zapewnić identyfikację udziału różnych sygnatur mutacji dla pojedynczej próbki guza. Ponadto serwer MutaGene zapewnia specyficzne dla mutagenu lub raka modele tła mutacji i sygnatury, które można zastosować do obliczenia oczekiwanej mutacji miejsca DNA i białka w celu oddzielenia względnego wkładu mutagenezy i selekcji w karcynogenezie.
Syntetyczna śmiertelność
Śmiertelność syntetyczna powstaje, gdy połączenie niedoborów ekspresji dwóch lub więcej genów prowadzi do śmierci komórki, podczas gdy niedobór tylko jednego z tych genów nie. Niedobory mogą powstać w wyniku mutacji, zmian epigenetycznych lub inhibitorów jednego z genów.
Potencjał terapeutyczny syntetycznej śmiertelności jako skutecznej strategii przeciwnowotworowej stale się poprawia. Ostatnio zastosowanie syntetycznej śmiertelności do ukierunkowanej terapii przeciwnowotworowej wzrosło ze względu na ostatnie prace naukowców, w tym Ronalda A. DePinho i współpracowników, dotyczące tego, co określa się mianem „ ubezpieczonej śmiertelności”. Muller i in. odkryli, że geny pasażera, znajdujące się w pobliżu chromosomów w stosunku do genów supresorowych nowotworu, są w niektórych nowotworach kolateralnie usuwane. Tak więc identyfikacja zbędnych genów z delecją boczną, pełniących istotną funkcję komórkową, może być niewykorzystanym rezerwuarem dla dalszego podejścia do syntetycznej letalności . Śmiertelność poboczna ma zatem ogromny potencjał w identyfikacji nowych i selektywnych celów terapeutycznych w onkologii. W 2012 roku Muller i in. zidentyfikowali, że homozygotyczna delecja zbędnego, niezbędnego glikolitycznego genu ENO1 w ludzkim glejaku (GBM) jest konsekwencją bliskości delecji locus supresora guza 1p36 i może mieć potencjał do syntetycznego podejścia do hamowania GBM. ENO1 jest jednym z trzech homologicznych genów ( EN02 , ENO3 ), który koduje enzym alfa-enolazę ssaków . ENO2 , który koduje enolazę 2 , ulega głównie ekspresji w tkankach nerwowych, co prowadzi do postulatu, że w GBM z delecją ENO1 ENO2 może być idealnym celem jako nadmiarowy homolog ENO1 . Muller odkrył, że zarówno genetyczne, jak i farmakologiczne hamowanie ENO2 w komórkach GBM z homozygotyczną delecją ENO1 wywołuje efekt syntetycznej śmiertelności poprzez selektywne zabijanie komórek GBM. W 2016 r. Muller i współpracownicy odkryli antybiotyk SF2312 jako bardzo silny inhibitor enolazy o zakresie nanomolarnym , który preferencyjnie hamuje proliferację komórek glejaka i przepływ glikolityczny w komórkach z delecją ENO1. SF2312 okazał się bardziej skuteczny niż pan-enolazy inhibitora PhAh i mają większą specyficzność ENO2 hamowanie przez ENO1 . Późniejsze prace tego samego zespołu wykazały, że to samo podejście można zastosować w przypadku raka trzustki , gdzie homozygotycznie usunięta SMAD4 powoduje delecję oboczną mitochondrialnego enzymu jabłkowego 2 ( ME2 ), dekarboksylazy oksydacyjnej niezbędnej dla homeostazy redoks . Dey i in. pokazują, że genomowa delecja ME2 w komórkach gruczolakoraka przewodowego trzustki skutkuje wysokimi endogennymi reaktywnymi formami tlenu, zgodnymi z rakiem trzustki napędzanym przez KRAS , i zasadniczo stymuluje komórki zerowe ME2 do syntetycznej śmiertelności przez zubożenie nadmiarowej izoformy ME3 zależnej od NAD(P)+. Stwierdzono, że w skutkach zubożenia ME3 pośredniczy hamowanie syntezy nukleotydów de novo wynikającej z aktywacji AMPK i apoptozy mitochondrialnej za pośrednictwem ROS. Tymczasem Oike i in. wykazano uogólniania tej koncepcji kierowania nadmiarowe istotne geny w procesie innym niż metabolizmu mianowicie smarca4 i SMARCA2 podjednostki w przebudowy chromatyny SWI / SNF złożone.
Niektóre onkogeny są niezbędne do przeżycia wszystkich komórek (nie tylko komórek nowotworowych). Tak więc leki, które eliminują te onkogeny (a tym samym zabijają komórki rakowe) mogą również uszkadzać normalne komórki, wywołując poważne choroby. Jednak inne geny mogą być niezbędne dla komórek rakowych, ale nie dla zdrowych komórek.
Leczenie oparte na zasadzie syntetycznej śmiertelności przedłużyło przeżycie pacjentów z rakiem i daje nadzieję na przyszłe postępy w odwracaniu procesu karcynogenezy. Główny typ syntetycznej śmiertelności działa na defekt naprawy DNA, który często inicjuje raka i jest nadal obecny w komórkach nowotworowych. Poniżej podano kilka przykładów.
Ekspresja BRCA1 lub BRCA2 jest niewystarczająca w większości raków piersi i jajnika o wysokim stopniu złośliwości, zwykle z powodu epigenetycznej metylacji ich promotora lub represji epigenetycznej przez nadekspresjonowane mikroRNA (patrz artykuły BRCA1 i BRCA2 ). BRCA1 i BRCA2 są ważnymi składnikami głównego szlaku homologicznej rekombinacyjnej naprawy pęknięć dwuniciowych. Jeśli jeden lub drugi jest niedobór, zwiększa ryzyko raka, zwłaszcza raka piersi lub jajnika. Zapasowy szlak naprawy DNA dla niektórych uszkodzeń zwykle naprawianych przez BRCA1 i BRCA2 zależy od PARP1 . Tak więc wiele raków jajnika reaguje na zatwierdzone przez FDA leczenie inhibitorem PARP, powodując syntetyczną śmiertelność komórek rakowych z niedoborem BRCA1 lub BRCA2. To leczenie jest również oceniane pod kątem raka piersi i wielu innych nowotworów w badaniach klinicznych fazy III w 2016 roku.
Istnieją dwa szlaki homologicznej rekombinacyjnej naprawy pęknięć dwuniciowych. Główny szlak zależy od BRCA1 , PALB2 i BRCA2, podczas gdy szlak alternatywny zależy od RAD52. Badania przedkliniczne, obejmujące epigenetycznie zredukowane lub zmutowane komórki z niedoborem BRCA (w hodowli lub wstrzykiwane myszom), pokazują, że hamowanie RAD52 jest syntetycznie śmiertelne w przypadku niedoboru BRCA.
Mutacje w genach wykorzystywanych w naprawie niedopasowania DNA (MMR) powodują wysoki wskaźnik mutacji. W nowotworach takie częste kolejne mutacje często wytwarzają „nie-własne” antygeny immunogenne. W badaniu klinicznym fazy II na ludziach, obejmującym 41 pacjentów, oceniono jedno syntetyczne, śmiertelne podejście do guzów z defektami MMR lub bez nich. Produkt genu PD-1 zwykle hamuje cytotoksyczne odpowiedzi immunologiczne. Hamowanie tego genu umożliwia większą odpowiedź immunologiczną. Kiedy pacjenci z rakiem z defektem MMR w guzach byli eksponowani na inhibitor PD-1, 67% - 78% pacjentów doświadczyło przeżycia wolnego od progresji związanej z układem immunologicznym. Natomiast w przypadku pacjentów bez wadliwego MMR, dodanie inhibitora PD-1 generowało tylko 11% pacjentów z przeżyciem wolnym od progresji immunologicznej. Zatem hamowanie PD-1 jest przede wszystkim syntetycznie śmiertelne w przypadku defektów MMR.
ARID1A , modyfikator chromatyny, jest wymagany do łączenia niehomologicznych końców , głównego szlaku naprawczego dwuniciowych pęknięć w DNA, a także pełni rolę regulatora transkrypcji. Mutacje ARID1A są jedną z 12 najczęstszych mutacji kancerogennych. Mutację lub epigenetycznie obniżoną ekspresję ARID1A stwierdzono w 17 typach raka. Badania przedkliniczne na komórkach i myszach pokazują, że syntetyczna śmiertelność niedoboru ARID1A następuje poprzez hamowanie aktywności metylotransferazy EZH2 lub przez dodanie inhibitora kinazy dazatynibu.
Innym podejściem jest indywidualne wybicie każdego genu w genomie i obserwowanie jego wpływu na komórki normalne i rakowe. Jeśli nokaut nieistotnego genu ma niewielki lub żaden wpływ na zdrowe komórki, ale jest śmiertelny dla komórek rakowych zawierających zmutowany onkogen, wówczas ogólnoustrojowa supresja tłumionego genu może zniszczyć komórki rakowe, pozostawiając zdrowe względnie nieuszkodzone. Technika ta została wykorzystana do identyfikacji inhibitorów PARP-1 w leczeniu nowotworów związanych z BRCA1 / BRCA2 . W tym przypadku połączona obecność hamowania PARP-1 i mutacji związanych z rakiem w genach BRCA jest śmiertelna tylko dla komórek rakowych.
Bazy danych do badań nad rakiem
The Cancer Genome Project to inicjatywa mająca na celu zmapowanie wszystkich somatycznych mutacji w raku. Projekt systematycznie sekwencjonuje eksony i flankujące połączenia splicingowe genomów guzów pierwotnych i linii komórek rakowych. Oprogramowanie COSMIC wyświetla dane wygenerowane z tych eksperymentów. Do lutego 2008 r. CGP zidentyfikowało 4746 genów i 2985 mutacji w 1848 guzach.
Projekt Cancer Genome Anatomy Project zawiera informacje o badaniach nad genomami, transkryptomami i proteomami raka.
Progenetix to onkogenomiczna referencyjna baza danych, prezentująca dane cytogenetyczne i molekularno-cytogenetyczne dotyczące nowotworów.
Oncomine zebrał dane z profili transkryptomów raka.
Integracyjna baza danych onkogenomicznych IntOGen i zestawy danych Gitools integrują wielowymiarowe dane onkogenomiczne człowieka sklasyfikowane według typu nowotworu. Pierwsza wersja IntOGen skupiała się na roli deregulacji ekspresji genów i CNV w nowotworach. W późniejszej wersji podkreślono mutacyjne geny kierowcy raka w 28 typach nowotworów. Wszystkie wydania danych IntOGen są udostępniane w bazie danych IntOGen.
Międzynarodowy Cancer Genome Consortium to największy projekt w celu gromadzenia danych rak ludzkiego genomu. Dane są dostępne za pośrednictwem strony internetowej ICGC. Pakiet BioExpress® Oncology Suite zawiera dane dotyczące ekspresji genów z próbek guzów pierwotnych, przerzutowych i łagodnych oraz próbek prawidłowych, w tym dopasowanych kontroli sąsiednich. Pakiet zawiera próbki złośliwości hematologicznej wielu znanych nowotworów.
Konkretne bazy danych dla modelowych zwierząt obejmują bazę danych Retrovirus Tagged Cancer Gene Database (RTCGD), która zebrała badania nad mutagenezą insercyjną retrowirusów i transpozonów w nowotworach myszy.
Rodziny genów
Analiza mutacji całych rodzin genów wykazała, że geny z tej samej rodziny mają podobne funkcje, jak przewidują podobne sekwencje kodujące i domeny białkowe . Dwie takie klasy to rodzina kinaz , zaangażowana w dodawanie grup fosforanowych do białek oraz rodzina fosfataz , zaangażowana w usuwanie grup fosforanowych z białek. Rodziny te zostały najpierw zbadane ze względu na ich widoczną rolę w przekazywaniu komórkowych sygnałów wzrostu lub śmierci komórki. W szczególności ponad 50% raków jelita grubego niesie mutację w genie kinazy lub fosfatazy. Gen 3 kinaz fosfatydyloinozytolowych ( PIK3CA ) koduje kinazy lipidowe, które często zawierają mutacje w nowotworach jelita grubego, piersi, żołądka, płuc i różnych innych. Terapie lekowe mogą hamować PIK3CA. Innym przykładem jest gen BRAF , jeden z pierwszych zaangażowanych w czerniaki. BRAF koduje kinazę serynowo - treoninową zaangażowaną w szlak sygnałowy wzrostu RAS-RAF- MAPK . Mutacje w BRAF powodują konstytutywną fosforylację i aktywność w 59% czerniaków. Przed BRAF genetyczny mechanizm rozwoju czerniaka był nieznany i dlatego rokowania dla pacjentów były złe.
Mitochondrialny DNA
Mutacje mitochondrialnego DNA (mtDNA) są powiązane z powstawaniem nowotworów. Zidentyfikowano cztery typy mutacji mtDNA:
Mutacje punktowe
Mutacje punktowe zaobserwowano w kodującym i niekodującym regionie mtDNA zawartego w komórkach nowotworowych. U osób z rakiem pęcherza, głowy/szyi i płuc mutacje punktowe w regionie kodującym wykazują oznaki podobieństwa do siebie. Sugeruje to, że kiedy zdrowa komórka przekształca się w komórkę nowotworową (transformacja nowotworowa), mitochondria wydają się być homogenne. Liczne mutacje punktowe zlokalizowane w niekodującym regionie D-pętli mitochondriów nowotworowych sugerują, że mutacje w tym regionie mogą być ważną cechą niektórych nowotworów.
Usunięcia
Ten typ mutacji jest sporadycznie wykrywany ze względu na mały rozmiar (<1kb). Pojawienie się pewnych specyficznych mutacji mtDNA (delecja 264 pz i 66 pz w genie ND1 podjednostki kompleksu 1) w wielu typach nowotworów dostarcza dowodów na to, że małe delecje mtDNA mogą pojawić się na początku nowotworzenia . Sugeruje to również, że ilość mitochondriów zawierających te delecje wzrasta wraz z postępem guza. Wyjątkiem jest stosunkowo duża delecja, która pojawia się w wielu nowotworach (znana jako „powszechna delecja”), ale więcej delecji mtDNA na dużą skalę znaleziono w normalnych komórkach w porównaniu z komórkami nowotworowymi. Może to wynikać z pozornie adaptacyjnego procesu komórek nowotworowych w celu wyeliminowania wszelkich mitochondriów, które zawierają delecje na dużą skalę („powszechna delecja” wynosi > 4 kb).
Wstawki
Dwie małe insercje mtDNA o wielkości ~260 i ~520 pz mogą być obecne w raku piersi, raku żołądka, raku wątrobowokomórkowym (HCC) i raku okrężnicy oraz w normalnych komórkach. Nie ustalono korelacji między tymi wstawkami a rakiem.
Skopiuj mutacje liczbowe
Charakterystyka mtDNA za pomocą testów reakcji łańcuchowej polimerazy w czasie rzeczywistym wykazuje obecność ilościowej zmiany liczby kopii mtDNA w wielu nowotworach. Oczekuje się, że wzrost liczby kopii nastąpi z powodu stresu oksydacyjnego. Z drugiej strony uważa się, że spadek jest spowodowany przez somatyczne mutacje punktowe w miejscu początku replikacji nici H i/lub homopolimerowy odcinek c D310 w regionie pętli D, mutacje w p53 (gen supresorowy guza) pośredniczy szlak i/lub nieefektywna aktywność enzymatyczna z powodu mutacji POLG . Każdy wzrost/spadek liczby kopii pozostaje wtedy stały w komórkach nowotworowych. Fakt, że ilość mtDNA jest stała w komórkach nowotworowych sugeruje, że ilość mtDNA jest kontrolowana przez znacznie bardziej skomplikowany system w komórkach nowotworowych, a nie po prostu zmieniana w wyniku nieprawidłowej proliferacji komórek. Rola zawartości mtDNA w ludzkich nowotworach jest najwyraźniej różna dla poszczególnych typów guzów lub miejsc.
| Rodzaj raka | Lokalizacja mutacji punktowych | Nukleotydowa pozycja delecji | Wzrost kopii mtDNA # | Zmniejszenie kopii mtDNA # | |||
|---|---|---|---|---|---|---|---|
| Pętla D | mRNA | tRNA | rRNA | ||||
| Pęcherz moczowy | x | x | x | 15,642-15,662 | |||
| Pierś | x | x | x | x | 8470-13,447 i 8482-13459 | x | |
| Głowa i szyja | x | x | x | x | 8470-13,447 i 8482-13459 | x | |
| Doustny | x | x | 8470-13,447 i 8482-13459 | ||||
| Rak wątrobowokomórkowy (HCC) | x | x | x | x | 306-556 i 3894-3960 | x | |
| przełykowy | x | x | x | 8470-13,447 i 8482-13459 | x | ||
| Żołądkowy | x | x | x | 298-348 | x | ||
| Prostata | x | x | 8470-13,447 i 8482-13459 | x | |||
57,7% (500/867) zawierało punkty somatyczne, a spośród 1172 zbadanych mutacji 37,8% (443/1127) znajdowało się w regionie kontrolnym pętli D, 13,1% (154/1172) znajdowało się w genach tRNA lub rRNA i 49,1% (575/1127) znaleziono w genach mRNA potrzebnych do wytwarzania kompleksów niezbędnych do oddychania mitochondrialnego.
Aplikacje diagnostyczne
Niektóre leki przeciwnowotworowe celują w mtDNA i wykazują pozytywne wyniki w zabijaniu komórek nowotworowych. W badaniach wykorzystano mutacje mitochondrialne jako biomarkery w terapii komórek rakowych. Łatwiej jest celować w mutację w mitochondrialnym DNA niż w jądrowym DNA, ponieważ genom mitochondrialny jest znacznie mniejszy i łatwiejszy do przeszukania pod kątem określonych mutacji. Zmiany zawartości mtDNA wykryte w próbkach krwi mogą służyć jako marker przesiewowy do przewidywania przyszłej podatności na raka, a także śledzenia progresji nowotworu złośliwego. Wraz z tymi potencjalnie pomocnymi właściwościami mtDNA nie jest on pod kontrolą cyklu komórkowego i jest ważny dla utrzymania wytwarzania ATP i homeostazy mitochondriów. Te cechy sprawiają, że celowanie w mtDNA jest praktyczną strategią terapeutyczną.
Biomarkery raka
Kilka biomarkerów może być przydatnych w określaniu stopnia zaawansowania, prognozowania i leczenia raka. Mogą one obejmować polimorfizmy pojedynczych nukleotydów (SNP), aberracje chromosomowe , zmiany liczby kopii DNA, niestabilność mikrosatelitarną, metylację regionu promotora , a nawet wysoki lub niski poziom białka.
Zobacz też
- Spersonalizowana onkogenomika
- Onkotektura
- Atlas genomu nowotworowego
- Międzynarodowe Konsorcjum Genomów Nowotworowych