Mediator (koaktywator) - Mediator (coactivator)
Mediator to wielobiałkowy kompleks, który działa jako koaktywator transkrypcji u wszystkich eukariontów . Została odkryta w 1990 roku w laboratorium Rogera D. Kornberga , laureata Nagrody Nobla z 2006 roku w dziedzinie chemii . Kompleksy mediatorowe oddziałują z czynnikami transkrypcyjnymi i polimerazą RNA II . Główną funkcją kompleksów mediatorowych jest przekazywanie sygnałów z czynników transkrypcyjnych do polimerazy.
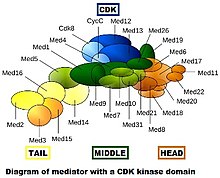
Kompleksy mediatorów są zmienne na poziomie ewolucyjnym, kompozycyjnym i konformacyjnym. Pierwszy obraz pokazuje tylko jedną „migawkę” tego, z czego może składać się dany kompleks mediatorowy, ale z pewnością nie przedstawia dokładnie konformacji kompleksu in vivo . Podczas ewolucji mediator stał się bardziej złożony. Uważa się, że drożdże Saccharomyces cerevisiae (prosty eukariota ) mają do 21 podjednostek w mediatorze rdzeniowym (z wyłączeniem modułu CDK), podczas gdy ssaki mają do 26.
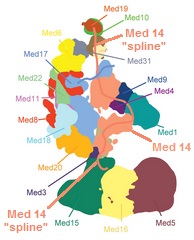
Poszczególne podjednostki mogą być nieobecne lub zastąpione innymi podjednostkami w różnych warunkach. Ponadto, w białkach mediatorowych istnieje wiele nieuporządkowanych regionów wewnętrznie , które mogą przyczyniać się do elastyczności konformacyjnej obserwowanej zarówno z, jak i bez innych związanych białek lub kompleksów białkowych. Bardziej realistyczny model kompleksu mediatora bez modułu CDK pokazano na drugim rysunku.
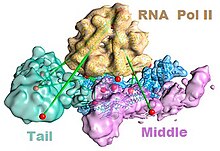
Kompleks mediatora jest niezbędny do udanej transkrypcji przez polimerazę RNA II. Wykazano , że mediator nawiązuje kontakt z polimerazą w kompleksie preinicjacji transkrypcji . Niedawny model pokazujący asocjację polimerazy z mediatorem pod nieobecność DNA pokazano na rycinie po lewej stronie. Oprócz polimerazy RNA II mediator musi również łączyć się z czynnikami transkrypcyjnymi i DNA. Model takich interakcji pokazano na rysunku po prawej stronie. Zauważ, że różne morfologie mediatora niekoniecznie oznaczają, że jeden z modeli jest poprawny; raczej te różnice mogą odzwierciedlać elastyczność mediatora, gdy oddziałuje on z innymi cząsteczkami. Na przykład, po związaniu wzmacniacza i promotora rdzenia, kompleks mediatora przechodzi zmianę składu, w której moduł kinazy dysocjuje od kompleksu, aby umożliwić połączenie z polimerazą RNA II i aktywację transkrypcji.
Kompleks Mediator znajduje się w jądrze komórkowym . Jest niezbędny do udanej transkrypcji prawie wszystkich promotorów genów klasy II u drożdży. Działa w ten sam sposób u ssaków. Funkcje mediatorów jak koaktywatorem i wiąże się z C-końcowej domeny od RNA polimerazy II holoenzymu , działając jako łącznik między tym enzymem i czynniki transkrypcyjne .
Struktura
Kompleks pośredniczący drożdży jest w przybliżeniu tak masywny jak mała podjednostka rybosomu eukariotycznego . Mediator drożdży składa się z 25 podjednostek, podczas gdy kompleksy mediatorów ssaków są nieco większe. Mediator można podzielić na 4 główne części: głowę, środek, ogon i przejściowo powiązany moduł kinazy CDK8.
Podjednostki mediatora mają wiele wewnętrznie nieuporządkowanych regionów zwanych „splajnami”, co może być ważne dla umożliwienia zmian strukturalnych mediatora, które zmieniają funkcję kompleksu. Rysunek pokazuje, w jaki sposób splajny podjednostki Med 14 łączą ze sobą dużą część kompleksu, jednocześnie zapewniając elastyczność.
Znaleziono lub wytworzono kompleksy mediatorów pozbawione podjednostki. Ci mniejsi mediatorzy mogą nadal normalnie funkcjonować w niektórych czynnościach, ale brakuje im innych zdolności. Wskazuje to na nieco niezależną funkcję niektórych podjednostek, będąc częścią większego kompleksu.
Inny przykład zmienności strukturalnej obserwuje się u kręgowców, u których 3 paralogi podjednostek modułu kinazy zależnej od cykliny wyewoluowały przez 3 niezależne zdarzenia duplikacji genów , po których następuje rozbieżność sekwencji.
Istnieje doniesienie, że mediator tworzy trwałe powiązania z określonym typem niekodującego RNA , ncRNA-a. Wykazano również , że te stabilne powiązania regulują ekspresję genów in vivo i zapobiegają im mutacje w MED12 , które wywołują zespół FG choroby u ludzi . W ten sposób struktura kompleksu mediatorowego może być wzmocniona przez RNA, jak również białkowe czynniki transkrypcyjne.
Funkcjonować
Mediator został pierwotnie odkryty, ponieważ był ważny dla funkcji polimerazy RNA II, ale pełni o wiele więcej funkcji niż tylko interakcje w miejscu startu transkrypcji.
Kompleks inicjacji rdzenia polimerazy RNA II-Mediator
Mediator jest kluczowym elementem inicjacji transkrypcji. Mediator oddziałuje z kompleksem przedinicjacyjnym, składającym się z polimerazy RNA II i ogólnych czynników transkrypcyjnych TFIIB, TFIID, TFIIE, TFIIF i TFIIH w celu stabilizacji i inicjacji transkrypcji. Badania kontaktów Mediator-RNA Pol II u pączkujących drożdży podkreśliły znaczenie kontaktów TFIIB-Mediator w tworzeniu kompleksu. Wykazano interakcje Mediatora z TFIID w kompleksie inicjacyjnym.
Wyjaśniono strukturę rdzeniowego mediatora (cMed), który jest związany z rdzeniowym kompleksem przedinicjacyjnym.
Synteza RNA
Kompleks preinicjacji, który zawiera mediator, czynniki transkrypcyjne, nukleosom i polimerazę RNA II, jest ważny dla pozycjonowania polimerazy na początku transkrypcji. Zanim nastąpi synteza RNA, polimeraza musi oddzielić się od mediatora. Wydaje się, że jest to osiągane przez fosforylację części polimerazy przez kinazę. Co ważne, mediator i czynniki transkrypcyjne nie oddzielają się od DNA w momencie rozpoczęcia transkrypcji przez polimerazę. Zamiast tego kompleks pozostaje na promotorze, aby rekrutować inną polimerazę RNA, aby rozpocząć kolejną rundę transkrypcji.
Istnieją pewne dowody sugerujące, że mediator w drożdżach jest zaangażowany w regulację transkryptów tRNA polimerazy III RNA (Pol III) Na poparcie tych dowodów, niezależny raport wykazał specyficzny związek mediatora z Pol III w Saccharomyces cerevisiae . Autorzy ci opisali również specyficzne powiązania z polimerazą RNA I i białkami zaangażowanymi w wydłużanie transkrypcji i obróbkę RNA, potwierdzając inne dowody na udział mediatora w wydłużaniu i przetwarzaniu.
Organizacja chromatyny
Mediator bierze udział w „zapętlaniu” chromatyny , co zbliża odległe regiony chromosomu do bliższego fizycznego sąsiedztwa. W takie zapętlenie bierze udział wspomniany powyżej ncRNA-a. Wzmacniające RNA (eRNA) mogą działać podobnie.
Oprócz tworzenia pętli euchromatyny mediator wydaje się być zaangażowany w tworzenie lub utrzymywanie heterochromatyny w centromerach i telomerach .
Transdukcja sygnału
Sygnalizacja TGFβ na błonie komórkowej skutkuje 2 różnymi szlakami wewnątrzkomórkowymi . Jeden z nich jest uzależniony od MED15, a drugi jest niezależny od MED15. Zarówno w komórkach ludzkich i Caenorhabditis elegans MED15 uczestniczy w homeostazie lipidowej w szlaku udziałem SREBPs w modelu roślin Arabidopsis thaliana ortolog z MED15 wymagana jest sygnalizacja przez hormon roślinny kwas salicylowy, a MED25 jest wymagana do aktywacji transkrypcji jasmonianu i odpowiedzi sygnalizacji cienia. Dwa elementy modułu CDK (MED12 i MED13) są zaangażowane w szlak sygnalizacyjny Wnt MED23 jest zaangażowany w szlak RAS / MAPK/ERK Ten skrócony przegląd pokazuje wszechstronność poszczególnych podjednostek mediatora i prowadzi do koncepcji, że mediator jest punkt ścieżek sygnałowych.
Choroba ludzka
Dokonano przeglądu udziału mediatora w różnych chorobach człowieka. Ponieważ hamowanie jednej interakcji wywołującego chorobę szlaku sygnałowego z podjednostką mediatora może nie hamować ogólnej transkrypcji potrzebnej do prawidłowego funkcjonowania, podjednostki mediatora są atrakcyjnymi kandydatami na leki terapeutyczne.
Interakcje
Metoda wykorzystująca bardzo delikatną lizę komórek w drożdżach, a następnie ko- immunoprecypitację z przeciwciałem przeciwko podjednostce mediatora (Med 17) potwierdziła prawie wszystkie wcześniej zgłoszone lub przewidywane interakcje i ujawniła wiele wcześniej niepodejrzewanych specyficznych interakcji różnych białek z mediatorem.
ŚREDNI 1
Omówienie wszystkich podjednostek mediatora wykracza poza zakres tego artykułu, ale szczegóły jednej z podjednostek ilustrują rodzaje informacji, które mogą być zbierane dla innych podjednostek.
regulacja przez mikroRNA
MikroRNA biorą udział w regulacji ekspresji wielu białek. Med1 jest celowany przez miR-1, co jest ważne w regulacji genów w nowotworach. Supresji guza miR-137 reguluje również MED1.
Rozwój embrionalny myszy
Mutanty null umierają we wczesnym wieku ciążowym (dzień embrionalny 11.5). Badając hipomorficzne mutanty (które mogą przetrwać o 2 dni dłużej), stwierdzono, że wady łożyska były przede wszystkim śmiertelne i że występowały również wady rozwoju serca i wątroby, ale wiele innych narządów było w normie
Komórki i tkanki myszy
U myszy można wytworzyć warunkowe mutacje, które w określonym czasie wpływają tylko na określone komórki lub tkanki, dzięki czemu mysz może osiągnąć dorosłość i można badać fenotyp osoby dorosłej . W jednym przypadku stwierdzono, że MED1 uczestniczy w kontrolowaniu czasu wystąpienia mejozy u samców myszy. Warunkowe mutanty w keratynocytach wykazują różnice w gojeniu się ran skóry. Warunkowego mutanta myszy stwierdzono zmianie dentystyczny nabłonka do naskórka nabłonka, co spowodowało rosnąć włosy związane z siekaczy.
Skład podjednostki
Kompleks mediator składa się co najmniej 31 podjednostek we wszystkich organizmach eukariotycznych badane: MED1 , MED4 , MED6 , MED7 , MED8 , MED9 , MED10 , MED11 , MED12 , MED13 , MED13L, MED14 , MED15 , MED16 , MED17 , MED18 , MED19 , MED20 , MED21 , MED22 , MED23 , MED24 , MED25 , MED26 , MED27 , MED28 , MED29 , MED30 , MED31 , CCNC i CDK8 . Istnieją trzy specyficzne dla grzybów składniki, określane jako Med2 , Med3 i Med5 .
W podjednostek , tworząc co najmniej trzy strukturalnie różne podmoduły. Moduły głowy i środkowe oddziałują bezpośrednio z polimerazą RNA II, natomiast moduł wydłużonego ogona oddziałuje ze specyficznymi dla genu białkami regulatorowymi . Mediator zawierający moduł CDK8 jest mniej aktywny niż Mediator pozbawiony tego modułu we wspieraniu aktywacji transkrypcyjnej .
- Moduł głowicy zawiera: MED6, MED8, MED11, SRB4/MED17, SRB5/MED18, ROX3/MED19, SRB2/MED20 i SRB6/MED22.
- Moduł środkowy zawiera: MED1, MED4, NUT1/MED5, MED7, CSE2/MED9, NUT2/MED10, SRB7/MED21 oraz SOH1/MED31. CSE2/MED9 współdziała bezpośrednio z MED4.
- Moduł ogona zawiera: MED2, PGD1/MED3, RGR1/MED14, GAL11/MED15 oraz SIN4/MED16.
- Moduł CDK8 zawiera: MED12, MED13, CCNC i CDK8. Poszczególne preparaty kompleksu Mediator pozbawione jednej lub więcej odrębnych podjednostek były różnie określane jako ARC, CRSP, DRIP, PC2, SMCC i TRAP.
W innych gatunkach
Poniżej znajduje się porównanie międzygatunkowe podjednostek kompleksu mediatora.
| Podjednostka nr | Ludzki gen | C. elegans gen | D. gen melanogaster | Gen S. cerevisiae | Sch. gen pombe |
|---|---|---|---|---|---|
| ŚREDNI 1 | ŚREDNI 1 | Sop3/mdt-1.1, 1.2 | ŚREDNI 1 | ŚREDNI 1 | med1 |
| Med2 | ŚREDNI 2 | ||||
| Med3 | PGD1 | ||||
| ŚREDNI 4 | ŚREDNI 4 | ŚREDNI 4 | ŚREDNI 4 | med4 | |
| Med5 | ORZECZ.1 | ||||
| ŚREDNI 6 | ŚREDNI 6 | MDT-6 | ŚREDNI 6 | ŚREDNI 6 | med6 |
| MED7 | MED7 | MDT-7/let-49 | MED7 | MED7 | med7 |
| MED8 | MED8 | MDT-8 | MED8 | MED8 | med8 |
| MED9 | MED9 | MED9 | CSE2 | ||
| MED10 | MED10 | MDT-10 | ORZECZ 2 | med10 | |
| MED11 | MED11 | MDT-11 | MED11 | MED11 | med11 |
| MED12 | MED12 | MDT-12/dpy-22 | MED12 | SRB8 | srb8 |
| MED12L | MED12L | ||||
| MED13 | MED13 | MDT-13/let-19 | MED13 | SSN2 | srb9 |
| ŚREDNIO 14 | ŚREDNIO 14 | MDT-14/rgr-1 | ŚREDNIO 14 | RGR1 | med14 |
| ŚREDNIO 15 | ŚREDNIO 15 | mdt-15 | ŚREDNIO 15 | GAL11 | YN91_SCHPO |
| ŚREDNI 16 | ŚREDNI 16 | ŚREDNI 16 | SIN4 | ||
| MED17 | MED17 | MDT-17 | MED17 | SRB4 | med17 |
| MED18 | MED18 | MDT-18 | MED18 | SRB5 | med18 |
| MED19 | MED19 | MDT-19 | MED19 | ROX3 | med19 |
| MED20 | MED20 | MDT-20 | MED20 | SRB2 | med20 |
| MED21 | MED21 | MDT-21 | MED21 | SRB7 | med21 |
| MED22 | MED22 | MDT-22 | MED22 | SRB6 | med22 |
| MED23 | MED23 | MDT-23/sur-2 | MED23 | ||
| MED24 | MED24 | MED24 | |||
| ŚREDNIO 25 | ŚREDNIO 25 | ŚREDNIO 25 | |||
| MED26 | MED26 | MED26 | |||
| MED27 | MED27 | MED27 | med27 | ||
| ŚREDNI 28 | ŚREDNI 28 | ŚREDNI 28 | |||
| MED29 | MED29 | MDT-19 | MED29 | ||
| ŚREDNIO 30 | ŚREDNIO 30 | ŚREDNIO 30 | |||
| MED31 | MED31 | MDT-31 | MED31 | SOH1 | med31 |
| CCNC | CCNC | cic-1 | CycC | SSN8 | pch1 |
| CDK8 | CDK8 | cdk-8 | Cdk8 | SSN3 | srb10 |