Wirusoid - Virusoid
| Okrągłe satelitarne RNA | |
|---|---|
|
Klasyfikacja wirusów |
|
| Grupa nieformalna : | Satelitarne kwasy nukleinowe |
| Grupa nieformalna : | Okrągłe satelitarne RNA |
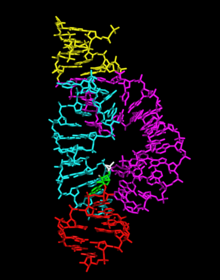
Wirusoidy to koliste jednoniciowe RNA (s) zależne od wirusów pod względem replikacji i enkapsydacji . Genom z virusoids składają kilkuset (200-400) nukleotydów i nie koduje dla wszystkich białek .
Virusoids są zasadniczo wiroidy , które zostały zamknięte w kapsułkach przez wirusa pomocnika białka płaszcza. Są zatem podobne do wiroidów pod względem sposobu replikacji (replikacja toczącego się koła ) i braku genów , ale różnią się tym, że wiroidy nie posiadają płaszcza białkowego. Zarówno wirusoidy, jak i wiroidy kodują rybozym typu hammerhead .
Wirusoidy, chociaż są badane w wirusologii , są raczej cząstkami subwirusowymi niż wirusami. Ponieważ są zależne od wirusów pomocniczych , są klasyfikowane jako satelity . Wirusoidy są wymienione w taksonomii wirusologicznej jako Satelity / Satelitarne kwasy nukleinowe / Podgrupa 3: Okrągły satelitarny RNA (s).
Definicja
W zależności od tego, czy stosowana jest definicja luźna, czy ścisła, termin wirusoid może również obejmować wirus zapalenia wątroby typu D (HDV). Podobnie jak wirusoidy roślinne, HDV jest kołowy, jednoniciowy i wspierany przez wirusa pomocniczego (wirusa zapalenia wątroby typu B ) w tworzeniu wirionów; jednak wiriony mają znacznie większy rozmiar genomu (~ 1700 nt) i kodują białko. Nie wykazują również podobieństwa sekwencji z grupą wirusoidów roślinnych.
Historia
Pierwszy wirusoid odkryto w roślinach Nicotiana velutina zakażonych wirusem pstrości tytoniu Velvet R2 (VTMOV). Te RNA są również określane jako RNA podobne do wiroidów, które mogą infekować ważne z komercyjnego punktu widzenia uprawy rolne i nie są samoreplikującymi się jednoniciowymi RNA. Replikacja RNA wirusoidów jest podobna do replikacji wiroidów, ale w przeciwieństwie do wiroidów, wirusoidy wymagają specyficznych wirusów „pomocniczych”.
Replikacja
Okrągła struktura cząsteczek wirusoidowego RNA jest idealna do replikacji toczącego się koła, w której wiele kopii genomu jest generowanych w wydajny sposób z pojedynczego zdarzenia inicjacji replikacji. Kolejną zaletą kolistych RNA jako pośredników replikacji jest to, że są one niedostępne i odporne na egzonukleazy. Dodatkowo ich wysoka zawartość GC i wysoki stopień samokomplementarności sprawiają, że są bardzo odporne na endonukleazy . Okrągłe RNA nakładają ograniczenia na fałdowanie RNA, przez co struktury drugorzędowe sprzyjające replikacji różnią się od tych zakładanych podczas samorozszczepiania za pośrednictwem rybozymu .
Roślinne satelitarne RNA i wirusoidy zależą od swoich odpowiednich wirusów pomocniczych pod względem replikacji, podczas gdy same wirusy pomocnicze są zależne od roślin w dostarczaniu niektórych składników wymaganych do replikacji. Dlatego złożona interakcja z udziałem wszystkich trzech głównych graczy, w tym satelitów, wirusów pomocniczych i roślin żywicielskich, jest niezbędna do replikacji satelitów/wirusoidów.
Wykazano, że replikacja satLTSV zachodzi poprzez mechanizm symetrycznego koła toczącego się, w którym satLTSV samorozszczepia nici (+) i (-). Stwierdzono, że obie nici (+) i (-) satLTSV są równie zakaźne. Niemniej jednak, ponieważ tylko nić (+) jest upakowana w cząstkach LTSV, zakłada się, że początek sekwencji składania (OAS) / struktura drugorzędowa występuje tylko na nici (+).
Gellatly i wsp., 2011 wykazali, że cała cząsteczka satLTSV ma znaczenie sekwencyjne i strukturalne, przy czym wszelkie mutacje (insercje/delecje) powodujące zakłócenie w ogólnej strukturze przypominającej pręciki cząsteczki wirusoidu są śmiertelne dla jej zakaźności. Obce nukleotydy wprowadzone do cząsteczki będą tolerowane tylko wtedy, gdy zachowają ogólną strukturę krzyżową satLTSV. Ponadto, wprowadzone obce sekwencje są eliminowane w kolejnych pokoleniach, aby ostatecznie odtworzyć satLTSV typu dzikiego.
Dlatego w RNA satLTSV cała sekwencja wydaje się być niezbędna do replikacji. Kontrastuje to z satRNA TBSV lub wadliwymi interferującymi RNA, w których tylko niewielka część ich odpowiednich sekwencji / struktur drugorzędowych okazała się wystarczająca do replikacji.
Rola struktur rybozymu w samorozszczepianiu i replikacji wirusoidów
Wirusoidy strukturalnie przypominają wiroidy, ponieważ posiadają natywne struktury drugorzędowe, które tworzą dwuniciowe cząsteczki podobne do pręcików z krótkimi gałęziami końcowymi. Zawierają również rybozymy typu hammerhead, które biorą udział w autokatalitycznym rozszczepianiu multimerów satRNA podczas replikacji toczącego się koła. Zaproponowano, że struktura rybozymu typu hammerhead w satLTSV jest tworzona tylko przejściowo, podobnie do obserwowanej przez Song i Millera (2004) z RNA satRPV ( serotyp RPV zbożowego żółtego karłowatości polerowirusa ). Ta struktura młotkowata zawiera krótki trzon III, który jest stabilizowany tylko dwoma nukleotydami o parze zasad. Ta niestabilna konformacja sugeruje zatem, że ma miejsce rozszczepianie w trybie podwójnego młotka. Struktury te są podobne do tych opisanych dla rybozymów CarSV i traszek, co sugeruje pradawny związek między tymi rozbieżnymi RNA. Obserwacja Collinsa i in., 1998, że dimer RNA satRYMV ulega samoodszczepieniu bardziej efektywnie niż monomer, jest zgodna z trybem cięcia z podwójną głową młota. Samorozszczepienie satRYMV w nici (+), a nie w nici (-) oznacza, że satRYMV replikuje się w asymetrycznym trybie replikacji toczącego się koła, podobnie jak w przypadku innych satelitów sobemowirusowych z wyjątkiem satLTSV.
Pochodzenie ewolucyjne
Biorąc pod uwagę takie właściwości, jak ich niewielki rozmiar, kolista struktura i obecność rybozymów , wiroidy mogły mieć starożytne pochodzenie ewolucyjne, różniące się od wirusów. Podobnie, brak jakiegokolwiek podobieństwa sekwencji między satelitarnymi RNA i ich wirusami-gospodarzami, roślinami-gospodarzami i wektorami owadzimi sugeruje, że te satelitarne RNA miały spontaniczne pochodzenie. Alternatywnie, siRNA i mikroRNA generowane podczas infekcji wirusowych mogą być amplifikowane przez replikacje wirusa pomocniczego, dzięki czemu cząsteczki te łączą się, tworząc satelitarne RNA.
Wirusoidy i wiroidy zostały porównane z okrągłymi intronami ze względu na podobieństwo ich wielkości. Zaproponowano, że wirusoidy i wiroidy pochodzą z intronów. Dokonano porównań między (-) nicią wiroidów a małą jądrową cząsteczką rybonukleoproteinową U1 ( snRNP ), co sugeruje, że wiroidy mogły uciec przed intronami. Dickson (1981) również zaobserwował takie homologie w obu niciach (+) i (-) wiroidów i wirusoidów. W szczególności virusoids wiroidy i wykazują kilka homologii strukturalnej i sekwencji do intronów grupy I, takie jak intron siebie splicingu z Tetrahymena thermophila .
Filogeneza oparta na ręcznym dopasowaniu z 2001 r. sugeruje, że wirusoidy mogą tworzyć własny klad jako siostrzana grupa do Avsunviroidae , które również posiadają rybozymy typu hammerhead. Jednak wspomniane wyrównanie nie jest dostępne, co utrudnia odtworzenie wyników .
Wirusoidy i inne okrągłe RNA to starożytne cząsteczki, które są badane z nowym zainteresowaniem. Wykazano, że okrągłe RNA posiadają szereg funkcji, począwszy od modulacji ekspresji genów, interakcji z białkami wiążącymi RNA (RBP) działającymi jak gąbki miRNA i są powiązane z wieloma chorobami człowieka, w tym starzeniem się i rakiem.
Rozwój
Abouhaidar i wsp., 2014 zademonstrowali jedyny przykład translacji białka i aktywności informacyjnego RNA w małym okrągłym satelickim RNA wirusa żółtej plamistości ryżu (scRYMV). Ta grupa zasugerowała, że scRYMV należy określić jako satelitarny RNA wirusa, który mógłby służyć jako system modelowy zarówno dla translacji, jak i replikacji.
Najbardziej obiecującym zastosowaniem tych czynników subwirusowych jest wytworzenie specyficznych wektorów, które można wykorzystać do przyszłego opracowania środków kontroli biologicznej chorób wirusowych roślin. System wektorowy można zastosować do nadekspresji i wyciszania obcych genów. Unikalnym przykładem obcego wektora ekspresyjnego jest satelitarny RNA wirusa mozaiki bambusa (satBaMV), który posiada otwartą ramkę odczytu, która koduje białko P20 o masie 20 kDa. Zaobserwowano, że gdy ten nieistotny region ORF został zastąpiony obcym genem, ekspresja obcego genu była wzmocniona lub nadeksprymowana. W przypadku wyciszania genów do inaktywacji specyficznej dla sekwencji można zastosować różne wektory satelitarne oparte na RNA. Satelitarny wirus mozaiki tytoniu (STMV) był pierwszym subwirusem, który został opracowany jako satelitarny system wyciszania wywołany wirusem (SVISS).