Ube3a-ATS - Ube3a-ATS
| SNHG14 | |||||||
|---|---|---|---|---|---|---|---|
| Identyfikatory | |||||||
| Skróty | SNHG14 , 115HG, LNCAT, NCRNA00214, UBE3A-AS, UBE3A-AS1, UBE3AATS, gen gospodarza małego jąderkowego RNA 14, IC-SNURF-SNRPN, U-UBE3A-ATS, UBE3A-ATS, Ube3a-ATS | ||||||
| Identyfikatory zewnętrzne | OMIM : 616259 Karty genowe : SNHG14 | ||||||
| Ortologi | |||||||
| Gatunek | Człowiek | Mysz | |||||
| Entrez |
|
||||||
| Zespół |
|
||||||
| UniProt |
|
||||||
| RefSeq (mRNA) |
|
|
|||||
| RefSeq (białko) |
|
|
|||||
| Lokalizacja (UCSC) | nie dotyczy | nie dotyczy | |||||
| Wyszukiwanie w PubMed | nie dotyczy | ||||||
| Wikidane | |||||||
| |||||||
UBE3A-ATS/Ube3a-ATS (człowiek/mysz), inaczej znany jako ligaza ubikwityny E3A-ATS, to nazwa antysensownej nici DNA, która jest transkrybowana jako część większego transkryptu zwanego LNCAT (duży niekodujący antysensowny transkrypt) w UBE3A locus. UBE3A locus nadruk i w centralnym układzie nerwowym wyraża się jedynie od allelu matki. Wyciszanie UBE3A na allelu ojcowskim uważa się wystąpić za pośrednictwem UBE3A-ATS części LNCAT , ponieważ niekodujących transkryptów antysensowne są często spotykane u nadrukiem loci. Delecja i/lub mutacja Ube3a na matczynym chromosomie powoduje zespół Angelmana (AS) i Ube3a-ATS mogą okazać się ważnym aspektem w znalezieniu terapii tej choroby. Podczas gdy u pacjentów z ZA matczyny allel Ube3a jest nieaktywny, allel ojcowski jest nienaruszony, ale epigenetycznie wyciszony. Niewyciszony allel ojcowski może być źródłem aktywnego białka Ube3a u pacjentów z ZA. Dlatego zrozumienie mechanizmów, w jaki Ube3a-ATS może być zaangażowany w wyciszanie ojcowskiego Ube3a, może prowadzić do nowych terapii na ZA. Możliwość tę wykazano w niedawnym badaniu, w którym lek topotekan podawany myszom cierpiącym na ZA, aktywował ekspresję ojcowskiego genu Ube3a poprzez obniżenie transkrypcji Ube3a-ATS .
Organizacja LNCAT
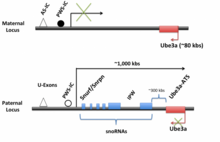
Ludzki UBE3A-ATS jest wyrażany jako część LNCAT głównie z ojcowskiego allelu w ośrodkowym układzie nerwowym (OUN). Transkrypt ma długość około 450 kb , zaczyna się od eksonów U i rozciąga się aż do UBE3A na przeciwległej nici, prawdopodobnie dalej. Promotor dla Snurf / Snrpn i środka tłoczenia można znaleźć w U-egzon regionu. Region promotora jest konieczny, ponieważ delecja tego obszaru znosi transkrypcję Ube3a-ATS . W pobliżu promotora znajduje się PWS-IC, a około 35 kb w górę od PWS-IC znajduje się AS-IC . Uważa się, że te dwa regiony kontrolują ekspresję całej nici LNCAT . Zaczynając od promotora, cały transkrypt może być transkrybowany, a po transkrypcji jest dalej przetwarzany i składany. Przeglądowi w Trends in Neurosci .
Obok regionu promotora eksonu U znajduje się Snrpn/Snurf, który może być alternatywnie składany w Snrpn lub Snurf u ludzi (u myszy pozostaje jako jeden transkrypt bicistronowy ). Snrpn koduje białko o nieznanej funkcji, które lokalizuje się w jądrze komórkowym . Snurf koduje małą jądrową rybonukleoproteinę . Chociaż większość tych białek bierze udział w splicingu , rola tego konkretnego białka nie jest jeszcze znana. Poniżej Snrpn/Snurf iw jego intronach znajdują się sekwencje kilku snoRNA C/D box . Większość snoRNA C/D box działa w metylacji innej niż mRNA . Jednak ostatnio stwierdzono, że jeden snoRNA na Ube3a-ATS , SNORD 115, zmienia alternatywny splicing pre-mRNA receptora serotoninowego 2C. Ponadto ten snoRNA ma zdolność do zmiany splicingu pięciu różnych mRNA. Wśród sekwencji snoRNA znajduje się zagnieżdżony IPW (imprinted Prader-Willi), niekodujący region, którego delecja, jak się uważa, powoduje zespół Pradera-Williego .
Systemy modelowe
Mysi i ludzki Ube3a-ATS/Ube3a są ortologiczne, a ogólna organizacja regionów jest podobna. Na przykład locus myszy zawiera również Snurf/Snrpn , snoRNA i IPW. Główne różnice to lokalizacja i długość transkryptów Ube3a-ATS . Ludzki Ube3a/Ube3a-ATS jest zlokalizowany na chromosomie 15 , podczas gdy mysi Ube3a jest zlokalizowany na chromosomie 7. Mysi LNCAT , w tym Ube3a-ATS , ma około 1000 kb długości, znacznie dłużej niż ludzki 450 kb LNCAT . Ze względu na podobną organizację myszy i człowieka LNCAT / UBE3A-TSA oraz fakt, że mysz UBE3A locus jest również nadruk , mysz to doskonały układ modelowy do badania imprintingu i interakcje między UBE3A / UBE3A-TSA . Ponadto neurony myszy nadal zachowują swój wzorzec imprintingu w kulturze.
Warianty i lokalizacje splotów
Chociaż cały transkrypt LNCAT , w tym transkrypt Ube3a-ATS, może podlegać transkrypcji, często jest on składany w celu włączenia i wykluczenia różnych eksonów. Różne warianty splotów są wyrażane w różnych typach tkanek i sytuacjach. W większości uważa się, że przynajmniej niektóre rodzaje Ube3a-ATS ulegają ekspresji w komórkach OUN, które są imprintowane, takich jak komórki Purkinjego i neurony hipokampa . Jednak istnieje regulacja czasoprzestrzenna zarówno dolnej, jak i górnej części tego transkryptu. i Journal of Neuroscience .
W embrionach myszy eksony Snurf/Snrpn wykryto w blastocystach około 7 dni po koitem i kontynuowano ich ekspresję przez cały okres rozwoju. W Snurf / Snrpn eksonów są ograniczone do tkanki CNS podczas rozwoju, a dopiero później, w okresie dorosłości ulegają ekspresji w innych tkankach. Egzonów Ube3a-ATS nie wykryto do 10 dni po koitem, a ich ekspresja była również ograniczona do OUN podczas rozwoju. Ogólnie rzecz biorąc, Ube3a-ATS jest wykrywany podczas początkowych etapów neurogenezy, podczas gdy Snurf/Snrpn ulega ekspresji w niezróżnicowanych komórkach prekursorowych iw trakcie różnicowania. Istnieje co najmniej 10 różnych izoform splicingowych według przeglądarki genomowej UCSC.
Według jednego z badań wariant splicingowy, który bezpośrednio zachodzi na Ube3a, znajduje się w cytoplazmie.
Zapobieganie ekspresji na obu allelach
Uważano, że specyficzny klaster imprinting center kontroluje zróżnicowaną ekspresję Ube3a-ATS na allelach matki i ojca . Istnieją dwa regiony w centrach nadruku (IC), które istnieją, związane z AS i PWS - AS-IC i PWS-IC. Te centra imprintingu są regionami kontrolnymi, które dyktują, czy otaczające geny i regiony mogą ulegać ekspresji i znajdują się zarówno w allelach matczynych, jak i ojcowskich. Podczas gdy zróżnicowane wzorce metylacji w genach matki i ojca są często związane z imprintingiem, AS-IC pozostaje niemetylowany w obu allelach. Jednak sąsiedni PWS-IC jest zmetylowany w allelu matczynym, ale pozostaje niemetylowany w allelu ojcowskim.
Podejrzewa się, że PWS-IC kontroluje ekspresję LNCAT i Ube3a-ATS . U myszy, u których usunięto PWS-IC, ekspresja Ube3a-ATS jest zmniejszona. W ośrodkowym układzie nerwowym Ube3a-ATS jest preferencyjnie wyrażany z ojcowskiego allelu, w którym PWS-IC nie jest metylowany. Z drugiej strony, w matczynym allelu, gdzie PWS-IC jest metylowany, Ube3a-ATS nie ulega ekspresji, co sugeruje, że metylacja PWS-IC w jakiś sposób zapobiega ekspresji Ube3a-ATS . Potwierdza to kilka badań, w których zapobieganie metylacji PWS-IC poprzez wybijanie transferaz metylowych w embrionalnych komórkach macierzystych skutkuje bialeliczną ekspresją Ube3a-ATS i wyciszeniem Ube3a w matczynym allelu.
Jednak metylacja nie jest jedynym procesem zaangażowanym w zapobieganie ekspresji matczynego Ube3a-ATS . Oczekuje się, że wyciśnięciu domeny interakcji z innymi białkami, które przyczyniają się do wyciszania z LNCAT i UBE3A-TSA w allelu matki. Na przykład, gdy MECP2 jest znokautowany, tak jak u pacjentów z zespołem Retta , Ube3a-ATS ulega biallelicznej ekspresji, zmniejszając ekspresję Ube3a z matczynego allelu.
Model kolizji
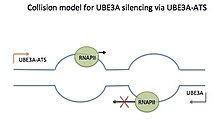
Obecnie istnieją trzy modele, które wyjaśniają, w jaki sposób Ube3a-ATS z LNCAT wycisza ojcowskiego Ube3a - model kolizji, model interakcji RNA-DNA i model interferencji dwuniciowego RNA . Chociaż modeli tych nie wykazano bezpośrednio dla Ube3a/Ube3a-ATS , uważa się je za wiarygodne na podstawie dowodów na wyciszanie innych naturalnych transkryptów antysensownych tymi metodami. Jednak model kolizji, ze względu na najnowsze badania pomocnicze, wydaje się najbardziej prawdopodobny.
Model kolizji można traktować jako drogę wystarczająco szeroką dla jednego samochodu. Inteligentny samochód jedzie z jednego kierunku, a pług z drugiego, aż w końcu się zderzy. Po zderzeniu pług popycha smarta do tyłu, kontynuując jazdę do przodu. W modelu kolizję dla UBE3A / UBE3A-TSA , polimerazy RNA (RNAPs) podróży w stosunku do siebie wzdłuż sensownych i antysensownych szablonów podczas transkrypcji. Szablony sensu i antysensu nakładają się na Ube3a i Ube3a-ATS . Dwie bańki transkrypcyjne zderzą się czołowo, a RNAP transkrypujący Ube3a-ATS , będący pługiem, wypchnie RNAP transkrybujący Ube3a (inteligentny samochód) do tyłu i ostatecznie poza szablon. Zapobiega to pełnej transkrypcji Ube3a.
Poparcie dla tego modelu pochodzi z dwóch ostatnich badań. Pierwsze badanie dotyczyło transkrypcji genów na niciach sensownych, na które nakładały się geny ulegające ekspresji na nici antysensownej . Im dłuższy region nakładania się, tym mniej wydajna była transkrypcja nici sensownej, co wskazuje, że transkrypcja jednej nici zakłóca transkrypcję drugiej nici. W innym badaniu bezpośrednio monitorowano kolizje między RNAPs transkrybując szablon za pomocą mikroskopii sił atomowych. RNAP zostały zatrzymane na fragmentach DNA i zderzyły się z innymi wydłużającymi się RNAP. Obrazy ukazywały przeciąganie dwóch RNAP bezpośrednio po zderzeniu, a także cofanie się jednego z RNAP.
Chociaż te badania nie zostały przeprowadzone dla Ube3a/Ube3a-ATS , zastosowanie mikroskopii sił atomowych do monitorowania transkrypcji w tym locus może zapewnić wgląd w to, w jaki sposób Ube3a jest faktycznie wyciszany przez Ube3a-ATS . Dalsze badania są nadal bardzo potrzebne, aby potwierdzić te modele dla Ube3a .
Sprzeczne badania
Podczas gdy kilka badań potwierdza pogląd, że Ube3a-ATS może być zaangażowany w ojcowskie wyciszanie Ube3a , inne badania temu zaprzeczają. W jednym badaniu, w szczególności przemawia przeciwko in cis wyciszania z Ube3 A przez UBE3A-TSA . W tym badaniu, gdy matczyny allel Ube3a został usunięty, zaobserwowano wzrost ojcowskiej ekspresji Ube3a-ATS . Sugeruje to, że zamiast ojcowskiego Ube3a-ATS kontrolującego ojcowskiego Ube3a , matczyny Ube3a w jakiś sposób tłumi ekspresję ojcowskiego Ube3a-AT , prawdopodobnie w trans zamiast w cis . Interakcję między matczynymi i ojcowskimi regionami homologicznymi tych genów faktycznie zaobserwowano w komórkach ludzkich i mysich podczas interfazy.
Jeden z mechanizmów, który można wyjaśnić w przypadku wyciszania trans, obejmuje interakcję między ojcowskim RNA Ube3a-ATS a matczynym mRNA Ube3a . Możliwe, że mRNA matki Ube3a oddziałuje z ojcowskim RNA Ube3a-ATS i zmniejsza stabilność obu tych transkryptów. Gdy tylko Ube3a-ATS jest tworzony bez Ube3a , Ube3a-ATS staje się bardziej stabilny.
Inne badanie sugeruje, że ekspresja Ube3a-ATS nie występuje w regionach z nadrukiem. Hybrydyzacja in situ nie ujawniła Ube3a-ATS w komórkach Purkinjego ani neuronach hipokampa. Jednak inne eksony poprzedzające, które odpowiadają Snurf / Snrpn, zostały wyrażone, co wskazuje, że model kolizji może nadal występować. Dlatego nadal potrzebne są dalsze badania.
Przyszłość
W kilku badaniach próbowano wykorzystać możliwość kontrolowania UBE3A ekspresji poprzez UBE3A-TSA . W AS ojcowski PWS-IC nie jest metylowany, co przypuszczalnie umożliwia ekspresję Ube3a-ATS . Dlatego też, gdyby metylacja PWS-IC była możliwa, transkrypcja Ube3a-ATS mogłaby zostać zabroniona, umożliwiając ekspresję Ube3a z allelu ojcowskiego, aby zrekompensować brak ekspresji z allelu matczynego. Przeprowadzono jednoroczne badanie z udziałem kilku pacjentów z AS. Pacjenci ci stosowali diety promujące metylację, które składały się z suplementów betainy , metafoliny , kreatyny i witaminy B12 . Jednak po roku wzorce metylacji u tych pacjentów nie uległy zmianie.
W innym badaniu przetestowano dużą bibliotekę różnych leków i zidentyfikowano kilka inhibitorów topoizomerazy I i II , które zwiększały ekspresję ojcowskiego Ube3a w neuronach myszy i myszy. Inhibitory topoizomerazy są szeroko stosowane jako chemioterapeutyki i powodują apoptozę komórek replikujących poprzez wywoływanie pęknięć dwuniciowych, które zatrzymują widełki replikacyjne . Jednak mechanizm ich działania w aktywacji ojcowskiego Ube3a nie jest jeszcze znany, ale może obejmować interferencję transkrypcyjną z Ube3a-ATS , ponieważ transkrypty Ube3a-ATS zmniejszyły się po leczeniu lekami. Grupa specjalnie wybrała do badania topotekan , który był najskuteczniejszy w niskim zakresie nanomolowym i jest już zatwierdzony przez Food and Drug Administration do leczenia kilku rodzajów nowotworów.