Protochlorofilid - Protochlorophyllide
|
|
| Nazwy | |
|---|---|
|
Nazwa IUPAC
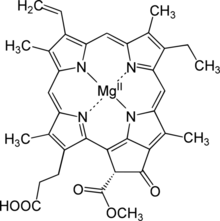
Magnez (21 R ) -3- (2-karboksyetylo) -14-etylo-21- (metoksykarbonylo) -4,8,13,18-tetrametylo-20-okso-9-winylo-3,4,23,25- tetradehydroforbino-23,25-diid
|
|
| Inne nazwy
Protochlorofilid monowinylu
|
|
| Identyfikatory | |
|
Model 3D ( JSmol )
|
|
| ChEBI | |
| ChemSpider | |
| KEGG | |
|
PubChem CID
|
|
|
|
|
|
| Nieruchomości | |
| C 35 H 32 MgN 4 O 5 | |
| Masa cząsteczkowa | 612,957 g / mol |
|
O ile nie zaznaczono inaczej, dane podano dla materiałów w ich stanie standardowym (przy 25 ° C [77 ° F], 100 kPa). |
|
| Referencje Infobox | |

Protochlorophyllide lub monowinylowy protochlorophyllide jest produktem pośrednim w biosyntezie z chlorofilu a . Brakuje fitolowego łańcucha bocznego chlorofilu i zredukowanego pirolu w pierścieniu D. Protochlorofilid jest silnie fluorescencyjny ; kumulujące się mutanty świecą na czerwono, jeśli są napromieniowane niebieskim światłem. U roślin okrytozalążkowych późniejsze etapy przekształcania protochlorofilidu w chlorofil są zależne od światła, a rośliny takie są blade ( chlorotyczne ), jeśli rosną w ciemności. Rośliny nagonasienne , algi i bakterie fotosyntetyzujące mają inny, niezależny od światła enzym i zielenieją również w ciemności.
Konwersja do chlorofilu
Enzymem, który przekształca protochlorofilid w chlorofilid a , kolejny związek pośredni na szlaku biosyntezy, jest reduktaza protochlorofilidu , EC 1.3.1.33. Istnieją dwa strukturalnie niepowiązane białka o tej aktywności: zależne od światła i ciemne operujące. Reduktaza zależna od światła potrzebuje światła do działania. Wersja ciemno operacyjna to zupełnie inne białko, składające się z trzech podjednostek, które wykazują znaczne podobieństwo sekwencji do trzech podjednostek azotazy , która katalizuje tworzenie się amoniaku z diazotu. Enzym ten może być starszy ewolucyjnie, ale (będąc podobnym do azotazy) jest bardzo wrażliwy na wolny tlen i nie działa, jeśli jego stężenie przekracza około 3%. Dlatego alternatywna, zależna od światła wersja musiała ewoluować.
Większość bakterii fotosyntetycznych ma reduktazy zarówno zależne od światła, jak i niezależne od światła. Rośliny okrytozalążkowe utraciły ciemną formę operacyjną i opierają się na 3 nieco różnych kopiach wersji zależnej od światła, często w skrócie POR A, B i C.Gonasienne mają znacznie więcej kopii podobnego genu ( sosna Loblolly ma około 11 Loblolly Pine ( Pinus taeda L.) Zawiera wiele eksprymowanych genów kodujących zależny od światła NADPH: oksydoreduktazę protochlorofilidu (POR) ). U roślin POR jest kodowany w jądrze komórkowym i dopiero później transportowany do miejsca pracy - chloroplastu . W przeciwieństwie do POR, w roślinach i algach, które mają enzym działający w ciemności, jest on przynajmniej częściowo kodowany w genomie chloroplastu .
Potencjalne zagrożenie dla rośliny
Sam chlorofil wiąże się z białkami i może przenosić zaabsorbowaną energię w wymaganym kierunku. Protochlorofilid występuje jednak głównie w postaci wolnej iw warunkach oświetleniowych działa jako fotouczulacz, tworząc silnie toksyczne wolne rodniki. Dlatego rośliny potrzebują skutecznego mechanizmu regulacji ilości prekursora chlorofilu. U roślin okrytozalążkowych odbywa się to na etapie kwasu δ-aminolewulinowego (ALA), jednego ze związków pośrednich na szlaku biosyntezy. Rośliny karmione ALA gromadzą wysokie i toksyczne poziomy protochlorofilidu, podobnie jak mutanty z zaburzonym systemem regulacyjnym.
Mutant Arabidopsis FLU z uszkodzoną regulacją może przeżyć tylko w ciągłej ciemności (protochlorofilid nie jest niebezpieczny w ciemności) lub w ciągłym świetle, kiedy roślina jest w stanie przekształcić cały wyprodukowany protochlorofil w chlorofil i nie kumuluje go nadmiernie pomimo braku rozporządzenie. U mutanta jęczmienia Tigrina (zmutowanego w tym samym genie) światło zabija większość tkanki liścia, która rozwinęła się w ciemności, ale część liścia, która powstała w ciągu dnia, przeżywa. W rezultacie liście pokryte są białymi paskami obszarów nekrotycznych, a liczba białych pasów jest zbliżona do wieku liścia w dniach. Obszary zielone przeżywają kolejne noce, prawdopodobnie dlatego, że synteza chlorofilu w dojrzałej tkance liści i tak jest znacznie zmniejszona.
Białko regulatorowe biosyntezy FLU
Pomimo wielu podejmowanych w przeszłości prób znalezienia mutanta, który w normalnych warunkach nadmiernie akumuluje protochlorofilid, obecnie (2009) znany jest tylko jeden taki gen ( grypa ). Grypa (po raz pierwszy opisana w) to kodowane w jądrze białko zlokalizowane w chloroplastach, które wydaje się zawierać tylko miejsca interakcji białko-białko. Obecnie nie wiadomo, które inne białka oddziałują za pośrednictwem tego łącznika. Białko regulatorowe jest białkiem transbłonowym, które znajduje się w błonie tylakoidów . Później odkryto, że znane od dawna mutanty Tigrina jęczmienia są również zmutowane w tym samym genie. Nie jest oczywiste, dlaczego nie zaobserwowano żadnych mutantów żadnego innego genu; być może mutacje w innych białkach zaangażowanych w łańcuch regulatorowy są śmiertelne. Grypa to pojedynczy gen, a nie członek rodziny genów .
Później, dzięki podobieństwu sekwencji, podobne białko znaleziono w glonach Chlamydomonas , co wskazuje, że ten podsystem regulacyjny istniał na długo przed utratą przez okrytozalążkowe niezależnego enzymu konwersji. W inny sposób białko regulatorowe Chlamydomonas jest bardziej złożone: jest większe, dwukrotnie przekracza błonę tylakoidów, zawiera więcej miejsc interakcji białko-białko, a nawet podlega alternatywnemu splicingowi . Wydaje się, że system regulacyjny uległ uproszczeniu w trakcie ewolucji.
Bibliografia
- ^ Wpis w bazie danych związków KEGG [1]
- ^ Willows, Robert D. (2003). „Biosynteza chlorofilów z protoporfiryny IX”. Raporty o produktach naturalnych . 20 (6): 327–341. doi : 10.1039 / B110549N . PMID 12828371 .
- ^ a b Meskauskiene R, Nater M, Goslings D, Kessler F, op den Camp R, Apel K. FLU: negatywny regulator biosyntezy chlorofilu u Arabidopsis thaliana. Materiały z National Academy of Sciences w Stanach Zjednoczonych Ameryki. 2001; 98 (22): 12826-31 pdf .
- ^ R. Caspi (2007-07-18). „3,8-diwinylo-chlorofilid a biosynteza I (tlenowa, zależna od światła)” . Baza danych MetaCyc Metabolic Pathway . Źródło 2020-06-04 . CS1 maint: zniechęcony parametr ( link )
- ^ KEGG wpis enzymu 1.3.1.33 [2]
- ^ Yuichi FujitaDagger i Carl E. Bauer (2000). Rekonstytucja niezależnej od światła reduktazy protochlorofilidu z oczyszczonych podjednostek Bchl i BchN-BchB. J. Biol. Chem., Vol. 275, wydanie 31, 23583-23588. [3]
- ^ S.Yamazaki, J.Nomata, Y. Fujita (2006) Różnicowe działanie podwójnych reduktaz protochlorofilidów dla biosyntezy chlorofilu w odpowiedzi na poziomy tlenu w środowisku w Cyanobacterium Leptolyngbya boryana . Plant Physiology, 2006, 142, 911-922 [4]
- ^ J Li, M. Goldschmidt-Clermont, poseł Timko (1997). ChlB kodowany w chloroplastach jest wymagany dla niezależnej od światła aktywności reduktazy protochlorofilidu u Chlamydomonas reinhardtii . Plant Cell 5 (12): 1817–1829. [5] .
- ^ a b Lee, Keun Pyo; Kim, Chanhong; Lee, Dae Won; Apel, Klaus (2003). „TIGRINA d, wymagana do regulacji biosyntezy tetrapiroli w jęczmieniu, jest ortologiem genu FLU z Arabidopsis thaliana ” . Listy FEBS . 553 (1–2): 119–124. doi : 10.1016 / s0014-5793 (03) 00983-9 . PMID 14550558 . S2CID 34038176 .
- ^ A Falciatore, L Merendino, F Barneche, M Ceol, R Meskauskiene, K Apel, JD Rochaix (2005). Białka FLP działają jako regulatory syntezy chlorofilu w odpowiedzi na sygnały świetlne i plastydowe u Chlamydomonas . Genes & Dev, 19: 176-187 [6]
