Akonitaza - Aconitase
| hydrataza togoniowata | |||||||||
|---|---|---|---|---|---|---|---|---|---|
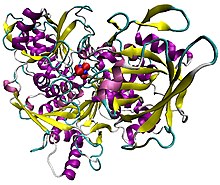 Ilustracja akonitazy świńskiej w kompleksie z klastrem [Fe 4 S 4 ]. Białko ma strukturę drugorzędową, atomy żelaza są niebieskie, a siarka czerwone.
| |||||||||
| Identyfikatory | |||||||||
| Nr WE | 4.2.1.3 | ||||||||
| Nr CAS | 9024-25-3 | ||||||||
| Bazy danych | |||||||||
| IntEnz | Widok IntEnz | ||||||||
| BRENDA | Wpis BRENDY | ||||||||
| ExPASy | Widok NiceZyme | ||||||||
| KEGG | Wpis KEGG | ||||||||
| MetaCyc | szlak metaboliczny | ||||||||
| PRIAM | profil | ||||||||
| Struktury WPB | RCSB PDB PDBe Suma PDB | ||||||||
| Ontologia genów | AmiGO / QuickGO | ||||||||
| |||||||||
| Rodzina akonitaz (hydrataza akonitatów ) | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 Struktura akonitazy.
| |||||||||
| Identyfikatory | |||||||||
| Symbol | Akonitaza | ||||||||
| Pfam | PF00330 | ||||||||
| InterPro | IPR001030 | ||||||||
| PROSITE | PDOC00423 | ||||||||
| SCOP2 | 1aco / zakres / SUPFAM | ||||||||
| |||||||||
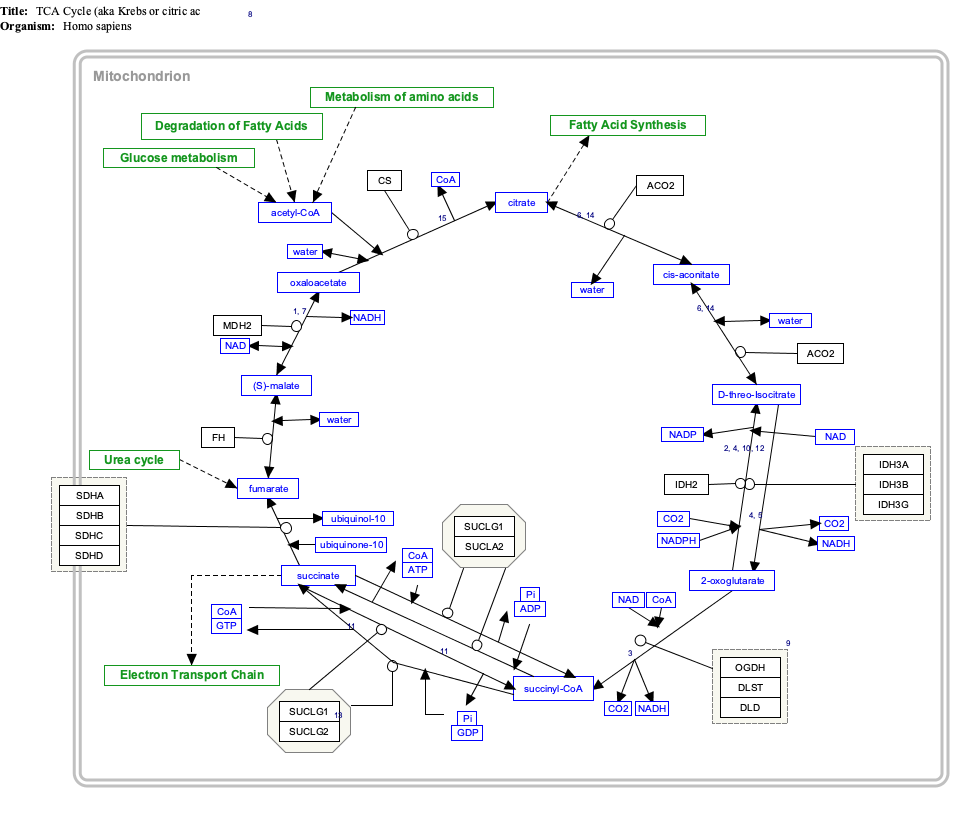
Akonitazy (akonitanową hydratazy, EC 4.2.1.3 ) jest enzymem, który katalizuje stereospecyficznego izomeryzacji z cytrynianem do izocytrynianową poprzez cis - akonitanową w cyklu kwasów trikarboksylowych , Nie- redoks -Active procesu.
Struktura
Akonitaza, wyświetlana w strukturach na prawym marginesie tej strony, ma dwie nieco różne struktury, w zależności od tego, czy jest aktywowana, czy nieaktywna. W formie nieaktywnej jego struktura podzielona jest na cztery domeny. Licząc od N-końca , tylko pierwsze trzy z tych domen są zaangażowane w bliskie interakcje z klastrem [3Fe-4S], ale miejsce aktywne składa się z reszt ze wszystkich czterech domen, w tym z większej domeny C-końcowej . Klaster Fe-S i anion SO 4 2- również znajdują się w centrum aktywnym. Kiedy enzym jest aktywowany, zyskuje dodatkowy atom żelaza, tworząc klaster [4Fe-4S]. Jednak struktura reszty enzymu jest prawie niezmieniona; konserwatywne atomy między tymi dwiema formami znajdują się zasadniczo w tych samych pozycjach, aż do różnicy 0,1 angstrema.
Funkcjonować
W przeciwieństwie do większości białek żelazowo-siarkowych, które działają jako nośniki elektronów, skupisko żelazowo-siarkowe akonitazy reaguje bezpośrednio z substratem enzymu. Akonitaza posiada klaster aktywny [Fe 4 S 4 ] 2+ , który może przekształcić się w formę nieaktywną [Fe 3 S 4 ] + . Wykazano, że trzy reszty cysteiny (Cys) są ligandami centrum [Fe 4 S 4 ]. W stanie aktywnym labilny jon żelaza klastra [Fe 4 S 4 ] nie jest koordynowany przez Cys, ale przez cząsteczki wody.
Element wiążący białko żelaza reaguje (IRE-BP) i 3-isopropylmalate dehydratazy (α-isopropylmalate izomeraza; EC 4.2.1.33 ), enzymu katalizującego drugi etap biosyntezy leucyny są znane akonitazy homologów. Żelazne elementy regulacyjne (IRE) stanowią rodzinę 28-nukleotydowych, niekodujących struktur typu łodyga-pętla, które regulują magazynowanie żelaza, syntezę hemu i wychwyt żelaza. Uczestniczą również w wiązaniu rybosomów i kontrolują obrót (degradację) mRNA . Specyficzne białko regulatorowe, IRE-BP, wiąże się z IRE w obu regionach 5' i 3', ale tylko z RNA w formie apo, bez klastra Fe-S. Ekspresja IRE-BP w hodowanych komórkach wykazała, że białko działa albo jako aktywna akonitaza, gdy komórki są nasycone żelazem, albo jako aktywne białko wiążące RNA, gdy komórki są zubożone w żelazo. Zmutowane białka IRE-BP, w których dowolna lub wszystkie trzy reszty Cys biorące udział w tworzeniu Fe-S są zastąpione seryną , nie wykazują aktywności akonitazy, ale zachowują właściwości wiązania RNA.
Akonitaza jest hamowana przez fluorooctan , dlatego fluorooctan jest trujący. Fluorooctan w cyklu kwasu cytrynowego może niewinnie wejść jako fluorocytrynian. Jednak akonitaza nie może wiązać tego substratu, a zatem cykl kwasu cytrynowego zostaje zatrzymany. Klaster żelaza i siarki jest bardzo wrażliwy na utlenianie nadtlenkiem .
Mechanizm
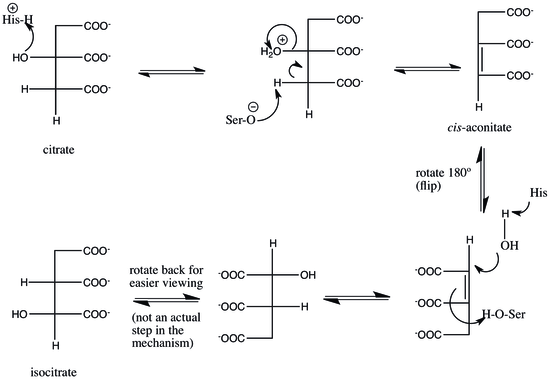
Akonitaza wykorzystuje mechanizm odwodnienia-hydratacji. Zaangażowane reszty katalityczne to His-101 i Ser-642. His-101 protonowanie grupa hydroksylowa C3 cytrynianu, co pozwala, aby pozostawić jak woda, i Ser-642 równolegle abstracts proton na węglu C2 tworzeniu wiązania podwójnego pomiędzy C2 i C3, i tworzące tak zwane cis -aconitate pośredni ( dwie grupy karboksylowe na podwójnym wiązaniu to cis ). Atom węgla, z którego usuwany jest wodór, to ten, który pochodzi ze szczawiooctanu w poprzednim etapie cyklu kwasu cytrynowego, a nie ten, który pochodzi z acetylo-CoA , mimo że te dwa węgle są równoważne, z wyjątkiem tego, że jeden jest „ pro- R " i inne " pro - S " ( patrz Prochiralność ). W tym momencie półprodukt jest obracany o 180°. Ten obrót jest określany jako „przerzucenie”. Z powodu tego odwrócenia mówi się, że półprodukt przechodzi z „trybu cytrynianowego” do „trybu izocytrynianowego”.
Jak dokładnie następuje ta klapka, jest dyskusyjne. Jedna z teorii głosi, że w ograniczającym szybkość etapie mechanizmu, cis- akonitynian jest uwalniany z enzymu, a następnie ponownie przyłączany w trybie izocytrynianowym, aby zakończyć reakcję. Ten etap ograniczający szybkość zapewnia, że w produkcie końcowym powstaje właściwa stereochemia , w szczególności (2R,3S). Inna hipoteza głosi, że cis- akonitynian pozostaje związany z enzymem, gdy przechodzi z trybu cytrynianowego do izocytrynianowego.
W każdym przypadku odwrócenie cis- akonitatu umożliwia zajście etapów odwodnienia i uwodnienia na przeciwległych powierzchniach półproduktu. Akonitaza katalizuje trans eliminację/dodawanie wody, a flip gwarantuje powstanie prawidłowej stereochemii w produkcie. Aby zakończyć reakcję, reszty seryny i histydyny odwracają swoje pierwotne działania katalityczne: histydyna, teraz zasadowa, odbiera proton z wody, przygotowując go jako nukleofil do ataku na C2, a protonowana seryna jest deprotonowana przez podwójny cis- akonitat wiązanie, aby zakończyć nawodnienie, tworząc izocytrynian.
Członkowie rodziny
Akonitazy są wyrażane w bakteriach u ludzi. Ludzie wyrażają następujące dwa izoenzymy akonitazy :
|
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Interaktywna mapa ścieżek
Kliknij poniżej geny, białka i metabolity, aby połączyć się z odpowiednimi artykułami.
Bibliografia
Dalsza lektura
- Frishman D, Hentze MW (lipiec 1996). „Zachowanie reszt akonitazy ujawnione przez wielokrotną analizę sekwencji. Implikacje dla zależności struktura/funkcja” . European Journal of Biochemistry / FEBS . 239 (1): 197–200. doi : 10.1111/j.1432-1033.1996.0197u.x . PMID 8706708 .
Linki zewnętrzne
- Akonitaza w Narodowej Bibliotece Medycznej USA Medical Subject Headings (MeSH)
- Proteopedia Aconitase - struktura Aconitase w interaktywnym 3D