FAM214A - FAM214A
| FAM214A | |||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Identyfikatory | |||||||||||||||||||||||||
| Skróty | FAM214A , KIAA1370, rodzina o podobieństwie sekwencji 214 członek A | ||||||||||||||||||||||||
| Identyfikatory zewnętrzne | MGI: 2387648 HomoloGene: 35065 GeneCards: FAM214A | ||||||||||||||||||||||||
| |||||||||||||||||||||||||
| |||||||||||||||||||||||||
| Ortologi | |||||||||||||||||||||||||
| Gatunki | Człowiek | Mysz | |||||||||||||||||||||||
| Entrez | |||||||||||||||||||||||||
| Ensembl | |||||||||||||||||||||||||
| UniProt | |||||||||||||||||||||||||
| RefSeq (mRNA) | |||||||||||||||||||||||||
| RefSeq (białko) | |||||||||||||||||||||||||
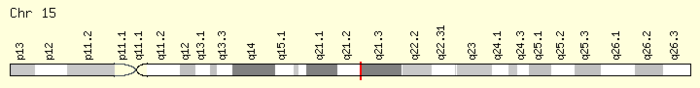
| Lokalizacja (UCSC) | Chr 15: 52,58 - 52,71 Mb | Chr 9: 74,95 - 75,03 Mb | |||||||||||||||||||||||
| Wyszukiwanie PubMed | |||||||||||||||||||||||||
| Wikidata | |||||||||||||||||||||||||
| |||||||||||||||||||||||||
Białko FAM214A , znane również jako rodzina białek o podobieństwie sekwencji 214, A (FAM214A) jest białkiem, które u ludzi jest kodowane przez gen FAM214A . FAM214A to gen o nieznanej funkcji znaleziony w locus q21.2-q21.3 na chromosomie 15 (ludzki) . Białko produkt tego genu dwie konserwowane domeny, jeden o nieznanej funkcji (DUF4210) i drugi o nazwie Chromosome_Seg. Chociaż funkcja białka FAM214A nie została scharakteryzowana, przewiduje się, że zarówno DUF4210, jak i Chromosome_Seg odgrywają rolę w segregacji chromosomów podczas mejozy.
Gen
Przegląd
FAM214A gen znajduje się na nici DNA ujemne (patrz sensowny (Molecular Biology) ) chromosomu 15 między pozycją 52,873,514 i 53,002,014; w ten sposób tworząc gen o długości 97,303 par zasad (pz). FAM214A był wcześniej oznaczony dwoma innymi aliasami, znanymi jako KIAA1370 i FLJ10980. Przewiduje się, że gen FAM214A zawiera 12 eksonów, które obejmują końcowy transkrypt mRNA o wielkości 4231 pz po wystąpieniu transkrypcji . To właśnie ten produkt mRNA ulega następnie translacji do końcowego białka FAM214A przy pomocy sekwencji promotora i czynników transkrypcyjnych . Promotor sekwencji mRNA FAM214A przewidziano i przeanalizowano za pomocą programu El Dorado na Genomatix. Ten promotor ma długość 601 par zasad i obejmuje część 5 'UTR.
Ekspresja genu
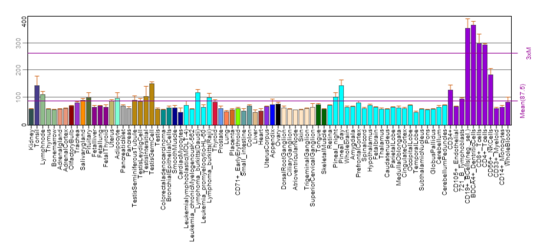
Według wielu źródeł, takich jak BioGPS i Atlas Ekspresji, FAM214A jest powszechnie (lub prawie tak blisko) wyrażany na niskich poziomach. Jak widać na poniższym obrazku BioGPS, istnieje znacznie wyższy poziom ekspresji w komórkach i tkankach związanych z odpornością, co sugeruje rolę immunologiczną; jednakże nie ma konkretnych dowodów in situ na poparcie tego twierdzenia. Dane dotyczące ekspresji zostały zebrane z szeregu badań przeprowadzonych na szerokim zakresie genów, dlatego niektóre dane są z natury sprzeczne.
Białko
Przegląd
Funkcja białka FAM214A u ludzi jest nadal nieznana; jednak istnieją trzy funkcjonalne powiązania terminów, w tym „proces biologiczny”, „składnik komórkowy” i funkcja molekularna ”, które opisują funkcję tego białka w Ontologii Genu, która przewiduje implikacje jego podstawowej funkcji in vivo . Produkt białkowy FAM214A składa się z 1076 aminokwasów (aa), przewiduje się, że ma masę cząsteczkową 121,700 daltonów i ma punkt izoelektryczny około pH 7,7. Przewiduje się, że białko to pozostanie w jądrze po transkrypcji na podstawie braku sekwencji peptydu sygnałowego i prognozy programu PSORTII. Ze względu na alternatywny splicing zaobserwowano dwie inne izoformy (Q32MH5-2 i Q32MH5-3). Różnią się one nieznacznie od produktu pierwotnego. Izoforma 2 ma cztery różne aminokwasy z zasad 960-960 i jest brakuje końca sekwencji z zasad 964-1076. Izoforma 3 ma siedem dodatkowych aminokwasów dodanych na początku sekwencji po metioninie.
Przewiduje się, że po translacji białko FAM214A pozostanie w jądrze przez więcej niż jeden typ podprogramu PSORT II. To białko ma sygnał pat4, jeden z dwóch „klasycznych” sygnałów lokalizacji jądrowej (NLS), zaczynając od reszty 709. Chociaż nie ma drugiego „klasycznego” NLS, pat7, ani „nieklasycznego” dwuczęściowego NLS to nadal przewiduje się, że będzie celem dla jądra na podstawie wyniku NCNN. Ten wynik przewiduje, na podstawie sekwencji aminokwasów, czy białko jest kierowane do jądra, czy do cytoplazmy. Dla białka FAM214A wynik NCNN przewidywał lokalizację jądra z 94,1% pewnością. W oparciu o te informacje PSORT generuje ogólne przewidywanie lokalizacji subkomórkowej białka. W przypadku FAM214A przewidywane wartości wynosiły 69,6% dla jądra w porównaniu z 13,0% dla mitochondriów, 8,7% dla cytoplazmy i 4,3% dla pęcherzyków wydzielniczych i retikulum endoplazmatycznego.
Modyfikacje potranslacyjne
Białko to najprawdopodobniej nie podlega znacznej liczbie modyfikacji potranslacyjnych z powodu braku sekwencji peptydu sygnałowego przewidzianej przez NetNGlyc i NetOGlyc na serwerze sieciowym ExPASy. Dzieje się tak, ponieważ wiele mechanizmów wewnątrzkomórkowych wykonujących modyfikacje potranslacyjne wymaga, aby białko przemieszczało się przez organelle, takie jak retikulum endoplazmatyczne i aparat Golgiego . Bez sekwencji peptydu sygnałowego białko na ogół nie opuszcza jądra, co zostało przewidziane przez PSORT II, jak opisano powyżej.
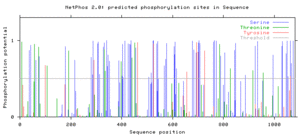
Analizę SAPS tego białka przeprowadzono w bazie danych swp23s.q, która wskazała na obecność nieprawidłowo dużej liczby aminokwasów seryny i nieprawidłowo małej liczby aminokwasów alaninowych w tym białku. Według artykułu przeglądowego Fayarda i wsp., Kinaza zależna od fosfoinozytydu 2 (PDK2) jest kinazą serynowo-treoninową, która jest ważna dla regulacji cyklu komórkowego. Ponieważ białko FAM214A ma większą liczbę grup seryny niż uważa się za normalne, istnieje możliwość, że PDK2 ma istotny wpływ na to białko. W celu ustalenia, czy w rzeczywistości przewidywano, że nadmierna liczba seryn jest fosforylowana, sekwencję białka przepuszczono przez program NetPhos z serwera internetowego ExPASy. Program ten przewidział fosforylację 69 seryn, 14 treonin i 9 tyrozyn. Zgodnie z powyższą analizą SAPS istnieje łącznie 134 seryny, co wskazuje, że przewiduje się, że około połowa jest fosforylowana in vivo . Schemat przewidywań fosforylacji pokazano po prawej stronie.
Inny rodzaj modyfikacji potranslacyjnych został przewidziany dla białka FAM214A w programie NetCorona na ExPASy. Program przewidział pojedyncze miejsce cięcia między pozycjami 214 i 215 w sekwencji białka FAM214A po translacji.
Interakcje białek
Istnieje wiele miejsc wiązania czynników transkrypcyjnych przewidzianych dla sekwencji promotora FAM214A. Kilka z nich o najwyższej przewidywanej ufności przedstawiono w poniższej tabeli.
Możliwe czynniki transkrypcji, które będą wiązać się z sekwencją promotora FAM214A
| Przewidywany współczynnik transkrypcji | Początek | Koniec | Pasmo | Pewność siebie |
| Element rozpoznający czynnik transkrypcyjny II B (TFIIB) | 97 | 103 | Negatywny | 1.0 |
| Białko szpikowego palca cynkowego MZF1 | 151 | 161 | Negatywny | 1.0 |
| Mielinowy czynnik transkrypcji 1, neuronalny czynnik C2HC z palcem cynkowym 1 | 388 | 400 | Negatywny | 0.945 |
| Miejsce wiązania receptora androgenu, miejsca IR3 | 495 | 513 | Negatywny | 0.923 |
| Wilms Tumor Suppressor | 1 | 17 | Pozytywny | 0.968 |
| Niepalindromiczne miejsca wiązania czynnika jądrowego I. | 27 | 47 | Pozytywny | 0.988 |
| Alternatywny wariant splicingu FOXP1, aktywowany w ESC | 383 | 383 | Pozytywny | 1.0 |
| Gen gruczolaka pleomorficznego 1 | 488 | 510 | Pozytywny | 1.0 |
| Gen 1 podobny do ETS (ELK-1) | 569 | 589 | Pozytywny | 0.961 |
Jedyne białko, co do którego przewiduje się, że według STRING będzie oddziaływać z białkiem FAM214A, nazywa się MFSD6L . Przewiduje się, że to białko należy do głównej nadrodziny pośredniczącej jako białko transbłonowe. Podobnie jak FAM214A, funkcja tego białka nie została jeszcze scharakteryzowana w drodze eksperymentów ani badań. Ponieważ to białko MFSD6L jest jedyną przewidzianą z jakąkolwiek pewnością interakcją białkową FAM214A, jego sekwencję przeprowadzono w programie PSORT II. Dane z podprogramu NLS przewidywały obecność jednej sekwencji pat4 i dwóch sekwencji NLS pat7, wskazując w ten sposób możliwą lokalizację jądrową. Z drugiej strony wynik NCNN przewidywał lokalizację cytoplazmatyczną z pewnością 94,1%, pozostawiając ogólny wynik PSORT II na poziomie 39,1% błony plazmatycznej, 39,1% retikulum endoplazmatycznego, 4,3% wakuolowej, 4,3% pęcherzyków układu wydzielniczego, 4,3% Golgiego, 4,3% mitochondrialne i 4,3% jądrowe. Jest to sprzeczne, ponieważ istnieją trzy całkowite sygnały lokalizacji jądrowej, ale może to wynikać z faktu, że znacząca transbłonowa natura białka MFSD6L może powodować problemy z tymi przewidywaniami.
Struktura drugorzędna i trzeciorzędowa
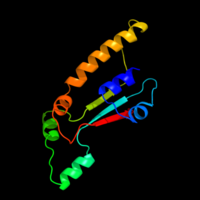
Struktura drugorzędowa białka FAM214A składa się z pewnej liczby alfa-helisy i beta arkuszy zgodnie z przewidywaniami i Biology Workbenchem P rotein H omology analogowo / Y R ecognition e ngine (PHYRE). Program PHYRE przewiduje, że 66 procent struktury drugorzędowej FAM214A jest nieuporządkowanych, a zatem nie można ich przeanalizować i przekształcić w przewidywaną strukturę trzeciorzędową. To było; jednak jest w stanie przewidzieć około 10 procent struktury białka z 95-procentowym znaczeniem. Schemat pokazany jest po lewej stronie.
Ochrona
Paralog
Pojedynczy gen paralogiczny został znaleziony na chromosomie 9 u Homo sapiens i został nazwany FAM214B (rodzina o podobieństwie sekwencji, B). FAM214B, chociaż uważany za paralog , ma znacząco inną sekwencję białka niż FAM214A. Kiedy te dwa zostały porównane ze sobą w BLAST NCBI, jedyne zaobserwowane znaczące podobieństwo dotyczyło ostatnich 200 aminokwasów (gdzie zlokalizowane są domeny DUF4210 i Chromosome_Seg). Chociaż podobieństwo między FAM214A i B jest niskie, te dwa białka należą do tej samej rodziny białek i zawierają te same dwie konserwowane domeny .
Ortologi
Białko FAM214A ma znaczną liczbę ortologów w dużej liczbie grup taksonomicznych, w tym ssaków , ptaków , reptilii , płazów , Actinopterygii , Echinoidea , Insecta , Trematoda , Crustacea , Tricoplacia, Anthozoa i Eurotiomycetes . Wskazuje to, że białko FAM214A jest dobrze konserwowane u eukariontów, ale nie wydaje się być konserwowane w bakteriach lub archeonach . We wszystkich ortologach najbardziej konserwowany region znajdował się blisko końca białka, w którym znajdują się konserwowane domeny (patrz poniżej). Ortologi dla ludzkiego białka FAM214A stwierdzono już w trufla czarnozarodnikowa , Talaromyces stipitatus i Aspergillus nidulans , które rozeszły około 1215000000 lat temu.
Ortologi dla białka FAM214A
| Rodzaj Gatunki | Nazwa zwyczajowa | Rozbieżność z ludzką linią (MYA) | Numer dostępu białka NCBI | Długość sekwencji | Procent identyczności z sekwencją ludzką | Wspólna nazwa genu |
| Homo sapiens | Człowiek | - | NP_062546.2 | 1076 | 100 | FAM214A |
| Pan troglodytes | Szympans zwyczajny | 6.3 | XP_003314724 | 1083 | 99 | FAM214A |
| Pan paniscus | Bonobo | 6.3 | XP_003827895.1 | 1076 | 100 | FAM214A |
| Rattus norvegicus | Szczur | 92.3 | NP_001100308 | 1074 | 100 | LOC300836 |
| Bos taurus | Krowa | 94.2 | XP_601152 | 1087 | 100 | KIAA1370 |
| Canus lupus familiaris | Pies | 94.2 | XP_544682 | 1081 | 100 | KIAA1370 |
| Ornithorhynchus anatinus | Dziobak | 167,4 | XP_001515207 | 1169 | 95 | KIAA1370 |
| Gallus gallus | kurczak | 296,0 | NP_001005811 | 1093 | 99 | FAM214A |
| Taeniopygia guttata | Zebra Finch | 296,0 | XP_002196177 | 1112 | 99 | FAM214A |
| Anolis carolinensis | Carolina Anole | 296,0 | XP_003227400 | 1086 | 99 | KIAA1370 |
| Xenopus tropicalis | Tropikalna żaba szponiasta | 371,2 | NP_001015702 | 946 | 98 | FAM214A |
| Danio rerio | Danio pręgowany | 400.1 | NP_001189349 | 1021 | 75 | FAM214A |
| Apis mellifera | Pszczoła miodna | 782,7 | XP_393903 | 1339 | 45 | LOC410423 |
| Strongylocentrotus purpuratus | Sea Urchin | 742,9 | XP_799179 | 297 | 27 | Podobne do FAM214A |
| muszka owocowa | Muszka owocowa | 782,7 | NP_610688 | 1297 | 27 | CG9005 |
| Schistosoma mansoni | Pasożyt Schistosome | 792,4 | XP_002579285 | 766 | 26 | Hipotetyczne białko |
| Daphnia pulex | Pchła wodna zwyczajna | 782,7 | EFX87516 | 200 | 18 | Hipotetyczne białko DAPPUDRAFT_207300 |
| Nematostella vectensis | Morski Anemon | 855,3 milionów lat temu | XP_001633540 | 191 | 18 | Hipotetyczne białko |
| Tuber melanosporum | Trufla | 1215,8 | XP_002841833 | 622 | 15 | Hipotetyczne białko |
| Talaromyces stipitatus | - | 1215,8 | XP_002478567 | 797 | 25 | Konserwatywne białko hipotetyczne |
| Aspergillus nidulans | Grzyb nitkowaty | 1215,8 | XP_658605 | 728 | 15 | hipotetyczne białko AN100 1.2 |
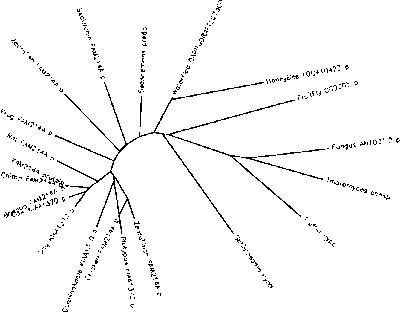
Filogeneza
Nieukorzenione drzewo filogenetyczne składające się z 20 ortologów zostało wygenerowane przez program CLUSTALW w Biology Workbench, aby zademonstrować ewolucyjny związek między FAM214A a jego ortologami.
Domeny chronione
W obrębie białka FAM214A znajdują się trzy dobrze konserwowane regiony. Obejmują one dobrze konserwowany region w pobliżu n-końca białka i dwie konserwowane domeny, w tym domenę o nieznanej funkcji 4210 (DUF4210) i domenę Chromosome_Seg w pobliżu c-końca . Schematyczny diagram tych trzech regionów przedstawiono poniżej. Nie przewiduje się, aby dobrze konserwowany region w pobliżu n-końca białka zawierał jakiekolwiek znane domeny lub motywy; jednakże miejsce rozszczepienia przewidziane przez NetCorona powyżej znajduje się w tym regionie i jest dobrze konserwowane w większości białek ortologicznych względem FAM214A. Dwie konserwatywne domeny znajdujące się na końcu tego białka są najważniejszą częścią peptydu opartą na historii ewolucji. Wszystkie organizmy w powyższej tabeli Ortholog z wyjątkiem dziobaka (któremu brakuje domeny Chromosome_Seg) zawierają obie te konserwatywne domeny w swojej sekwencji białkowej.