Zachowane indele podpisu - Conserved signature indels
Konserwatywne wstawki i delecje sygnatur ( CSI ) w sekwencjach białkowych stanowią ważną kategorię markerów molekularnych do zrozumienia zależności filogenetycznych. CSI, wywołane rzadkimi zmianami genetycznymi, dostarczają użytecznych markerów filogenetycznych, które mają na ogół określoną wielkość i są otoczone po obu stronach konserwatywnymi regionami, aby zapewnić ich wiarygodność. Podczas gdy indele mogą być arbitralnymi wstawkami lub delecjami, CSI definiuje się jako tylko te indele białka, które są obecne w konserwowanych regionach białka.
CSI, które są ograniczone do określonego kladu lub grupy gatunków, na ogół dostarczają dobrych markerów filogenetycznych wspólnego pochodzenia ewolucyjnego. Ze względu na rzadkość i wysoce specyficzny charakter takich zmian, jest mniej prawdopodobne, że mogą one powstać niezależnie w wyniku ewolucji zbieżnej lub równoległej (tj. homoplazji), a zatem prawdopodobnie reprezentują synapomorfię . Inne mylące czynniki, takie jak różnice w szybkości ewolucji w różnych miejscach lub między różnymi gatunkami, również generalnie nie mają wpływu na interpretację CSI. Określając obecność lub nieobecność CSI u gatunków z grupy obcej, można wywnioskować, czy pierwotna forma CSI była wstawką czy delecją i można to wykorzystać do rozwinięcia zakorzenionego związku filogenetycznego między organizmami.
Stwierdzono, że większość zidentyfikowanych CSI wykazuje wysoką wartość predykcyjną i generalnie zachowuje specyficzność dla pierwotnie zidentyfikowanych kladów gatunków. Dlatego na podstawie ich obecności lub braku powinno być możliwe zidentyfikowanie zarówno znanych, jak i wcześniej nieznanych gatunków należących do tych grup w różnych środowiskach.
Rodzaje
Specyficzne dla grupy
CSI specyficzne dla grupy są zwykle wspólne dla różnych gatunków należących do określonego taksonu (np. rodzaju, rodziny, klasy, rzędu, gromady), ale nie występują w innych grupach. Te CSI zostały najprawdopodobniej wprowadzone u przodka grupy gatunków, zanim członkowie taksonów się rozeszli. Dostarczają molekularnych środków do odróżnienia członków określonego taksonu od wszystkich innych organizmów.
Rysunek 1 pokazuje przykład 5aa CSI znalezionego we wszystkich gatunkach należących do taksonu X. Jest to charakterystyczna cecha tego taksonu, ponieważ nie występuje u żadnego innego gatunku. Sygnatura ta została prawdopodobnie wprowadzona u wspólnego przodka gatunku z tego taksonu. Podobnie inne sygnatury specyficzne dla grupy (nie pokazane) mogą być współdzielone przez A1 i A2 lub B1 i B2 itd., a nawet przez X1 i X2 lub przez X3 i X4 itd. Grupy A, B, C, D i X na tym schemacie może odpowiadać różnym typom bakteryjnym lub eukariotycznym .
W przeszłości stosowano CSI specyficzne dla grupy w celu określenia związku filogenetycznego wielu typów i podgrup bakteryjnych w ich obrębie. Na przykład 3-aminokwasowa wstawka była w unikalny sposób dzielona przez członków typu Thermotogae w niezbędnym rybosomalnym białku L7/L12 50S , w obrębie wysoce konserwatywnego regionu (82-124 aminokwasy). Nie występuje on w żadnym innym gatunku bakterii i można go wykorzystać do scharakteryzowania członków typu Thermotogae od wszystkich innych bakterii. Do scharakteryzowania podgrup w typie Thermotogae wykorzystano również CSI specyficzne dla grupy .
Wiele grup lub linia główna
Główne typy CSI to te, w których zachowana wstawka lub delecja jest wspólna dla kilku głównych typów, ale nieobecna w innych typach.
Rysunek 2 pokazuje przykład 5aa CSI znalezionego w konserwatywnym regionie, który jest powszechnie obecny w gatunkach należących do typu X, Y i Z, ale nie występuje w innych typach (A, B i C). Sygnatura ta wskazuje na specyficzny związek taksonów X, Y i Z, a także A, B i C. Na podstawie obecności lub braku takiego indelu w gatunkach z grupy obcej (np. Archaea) można wywnioskować, czy indel jest wstawką lub delecją i która z tych dwóch grup A, B, C lub X, Y, Z jest przodkiem.
Główne CSI były używane w przeszłości do określenia związku filogenetycznego wielu typów bakteryjnych. Duża CSI około 150-180 aminokwasów w obrębie konserwowanego regionu gyrazy B (między aminokwasami 529-751) jest powszechnie podzielonych między Proteobacteria , Chlamydiales , Planctomycetes i Aquificales gatunków. Ten CSI jest nieobecny w innych szczepach bakteryjnych przodków, jak również w Archaea . Podobnie duża CSI z około 100 aminokwasów, w rpoB homologów (między aminokwasami 919-1058) jest obecny w różnych gatunków należących do Proteobacteria , bacteroidetes-Chlorobi , Chlamydiales , Planctomycetes i Aquificales . Ten CSI jest nieobecny w innych szczepach bakteryjnych przodków, jak również w Archaea . W obu przypadkach można wywnioskować, że grupy pozbawione CSI są przodkami.
Badania ewolucyjne oparte na CSI
Kluczową kwestią w filogenezie bakterii jest zrozumienie, w jaki sposób różne gatunki bakterii są ze sobą powiązane i ich kolejność rozgałęzień od wspólnego przodka. Obecnie większość drzew filogenetycznych opiera się na 16S rRNA lub innych genach/białkach. Te drzewa nie zawsze są w stanie rozwiązać kluczowe pytania filogenetyczne z dużą dozą pewności. Jednak w ostatnich latach w tych poszukiwaniach pomogło odkrycie i analiza konserwatywnych indeli (CSI) w wielu powszechnie występujących białkach. Uważa się, że prowadzące do nich zdarzenia genetyczne miały miejsce w ważnych ewolucyjnych punktach rozgałęzień, a wzorce rozmieszczenia gatunków dostarczają cennych informacji na temat kolejności rozgałęzień i wzajemnych relacji między różnymi typami bakterii.
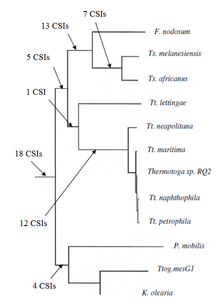
Termotogi
Ostatnio związek filogenetyczny grupy Thermotogae został scharakteryzowany w oparciu o podejście CSI. Wcześniej nie były znane żadne biochemiczne ani molekularne markery , które mogłyby wyraźnie odróżnić gatunek tego gromady od wszystkich innych bakterii. Odkryto ponad 60 CSI, które były specyficzne dla całej rodziny Thermotogae lub jej różnych podgrup. 18 CSI jest wyjątkowo obecnych w różnych gatunkach Thermotogae i dostarcza markerów molekularnych dla gromady. Dodatkowo było wiele CSI, które były specyficzne dla różnych podgrup termotogi. 12 CSI było specyficznych dla kladu składającego się z różnych gatunków Thermotoga z wyjątkiem Tt. Letingae. 14 CSI było specyficznych dla kladu składającego się z rodzajów Fervidobacterium i Thermosipho, a 18 CSI było specyficznych dla rodzaju Thermosiphon.
Wreszcie zgłoszono 16 CSI , które były wspólne dla niektórych lub wszystkich gatunków Thermotogae lub niektórych gatunków z innych taksonów , takich jak Archaea , Aquificae , Firmicutes , Proteobacteria , Deinococcus , Fusobacteria , Dictyoglomus , Chloroflexi i eukariota . Wspólna obecność niektórych z tych CSI może wynikać z bocznego transferu genów (LGT) między tymi grupami. Jednak liczba CSI, które są powszechnie dzielone z innymi taksonami, jest znacznie mniejsza niż te, które są specyficzne dla Thermotogae i nie wykazują żadnego specyficznego wzoru. Dlatego nie mają znaczącego wpływu na rozróżnienie Thermotogae.
Archea
Mezofilne Crenarchaeotes zostały niedawno umieszczone w nowym typie Archaea zwanym Thaumarchaeota . Jednak istnieje bardzo niewiele markerów molekularnych, które mogą odróżnić tę grupę archeonów od gromady Crenarchaeota. Przeprowadzono szczegółowe badanie filogenetyczne przy użyciu podejścia CSI w celu rozróżnienia tych typów pod względem molekularnym. W różnych Thaumarchaeota znaleziono 6 CSI, a mianowicie Cenarchaeum symbiosum , Nitrosopumilus maritimus i pewną liczbę niehodowanych morskich crenarchaeot. Stwierdzono 3 CSI, które były wspólne dla gatunków należących do Thaumarchaeota i Crenarchaeota. Ponadto znaleziono szereg CSI, które są specyficzne dla różnych rzędów Crenarchaeota – 3 CSI dla Sulfolobales , 5 CSI dla Thermoproteales , w końcu 2 CSI wspólne dla Sulfolobales i Desulfurococcales . Opisane sygnatury dostarczają nowych środków do rozróżniania Crenarchaeota i Thaumarchaeota, dodatkowo mogą być użyte jako narzędzie do klasyfikacji i identyfikacji gatunków pokrewnych.
Pasteurelle
Obecnie wyróżnia się członków rzędu Pasteurellales głównie na podstawie ich pozycji w rozgałęzieniu drzewa 16srRNA. Obecnie znanych jest bardzo niewiele markerów molekularnych, które potrafią odróżnić przedstawicieli tego rzędu od innych bakterii. Ostatnio zastosowano podejście CSI do wyjaśnienia filogenetycznych relacji między gatunkami w tej kolejności; odkryto ponad 40 CSI, które były w unikalny sposób wspólne dla wszystkich lub większości gatunków. W obrębie tego Pasteurellales powstają dwa główne klady: klad I, obejmujący Aggregatibacter , Pasteurella , Actinobacillus succinogenes, Mannheimia succiniciproducens, Haemophilus influenzae i Haemophilus somnus, był wspierany przez 13 CSI. Klad II, obejmujący Actinobacillus pleuropneumoniae, Actinobacillus minor, Haemophilus ducreyi , Mannheimia haemolytica i Haemophilus parasuis, był wspierany przez 9 CSI. Na podstawie tych wyników zaproponowano podział Pasteurellales z obecnej jednej rodziny na dwie różne. Dodatkowo opisane sygnatury zapewniłyby nowe sposoby identyfikacji nieodkrytych gatunków Pasteurellales.
Gammaproteobakterie
Klasa Gammaproteobacteria tworzy jedną z największych grup bakterii. Obecnie odróżnia ją od innych bakterii wyłącznie drzewa filogenetyczne oparte na 16s rRNA . Nie są znane żadne cechy molekularne charakterystyczne dla tej klasy lub jej różnych podgrup. Przeprowadzono szczegółowe badanie oparte na CSI, aby lepiej zrozumieć filogenezę tej klasy. W pierwszej kolejności stworzono drzewo filogenetyczne oparte na połączonych sekwencjach szeregu powszechnie występujących białek. Kolejność rozgałęzienia poszczególnych zleceń z klasy gammaproteobacteria (od najnowszych do najwcześniejszych rozbieżne) była następująca: Enterobacteriales > Pasteurellales > Vibrionales , Aeromonadales > Alteromonadales > Oceanospirillales , Pseudomonadales > Chromatiales , Legionellales , Methylococcales , Xanthomonadales , Cardiobacteriales , Thiotrichales . Dodatkowo odkryto 4 CSI, które były unikalne dla większości gatunków z klasy Gammaproteobacteria. Delecja 2 aminokwasów w transformylazie AICAR była w unikalny sposób wspólna dla wszystkich gammaproteobakterii z wyjątkiem Francisella tularensis . Delecję 4 aa w podjednostce b polimerazy RNA i delecję 1 aa w rybosomalnym białku L16 stwierdzono wyłącznie u różnych gatunków należących do rzędów Enterobacteriales , Pasteurellales , Vibrionales , Aeromonadales i Alteromonadales , ale nie znaleziono ich u innych gammaproteobakterii. Wreszcie, delecja 2 aa w syntetazie leucylo-tRNA była powszechnie obecna w powyższych rzędach klasy Gammaproteobacteria i u niektórych członków rzędu Oceanospirillales . W innym badaniu opartym na CSI zidentyfikowano również 4 CSI, które są wyłączne dla rzędu Xanthomonadales. Podsumowując, te dwa fakty pokazują, że Xanthomonadales jest grupą monofiletyczną, która jest przodkiem innych Gammaproteobacteria, co dodatkowo pokazuje, że Xanthomonadales jest niezależnym pododdziałem i stanowi jedną z najgłębiej rozgałęzionych linii w kladzie Gammaproteobacteria.
Grzyby
Dokładny związek filogenetyczny między roślinami , zwierzętami i grzybami nie jest dobrze poznany. Przeprowadzono małe badanie oparte na CSI w celu wyjaśnienia tej zależności. Do zestawienia zwierząt i grzybów jako grupy monofiletycznej i wykluczenia roślin wykorzystano cztery CSI. Te CSI znaleziono w dwóch podstawowych białkach komórkowych, czynniku elongacji I i enolazie . Jednak tradycyjnie ten specyficzny związek między grzybami a zwierzętami nie był wspierany.